Uso de PCR múltiple en el diagnóstico simultáneo de parasitosis
Simposio. Nuevos desarrollos en el diagnóstico de parásitos Laboratorio de Malaria y otras Parasitosis Emergentes, Servicio de Parasitología, Centro Nacional de Microbiología, Instituto de Salud Carlos III, Madrid, España Biomédica 2011;31(sup.3):3-315
Introducción
El primer problema de los servicios de salud frente a las enfermedades de tipo microbiológico es la realización de un diagnóstico sensible y eficaz en los primeros estadios de la enfermedad.
En general, los métodos clásicos de diagnóstico de las enfermedades infecciosas, especialmente en el campo de la parasitología, no están exentos de inconvenientes. Los métodos indirectos basados en la detección de anticuerpos en algunos casos no pueden diferenciar infecciones actuales de infecciones pasadas, además de ser frecuente que presenten reacciones cruzadas con otros patógenos. Los métodos directos, como cultivos o la microscopía son de efectividad relativa, debido al nivel de sensibilidad, a la complejidad de las técnicas de tinción y cultivo, a la falta de diferenciación morfológica de especies o a la subjetividad del observador. Por el contrario, los métodos directos basados en la detección del ADN del patógeno han mostrado una alta sensibilidad y especificidad, mejorando el índice de diagnósticos correctos con el beneficio para el paciente y para la sanidad pública disminuyendo el tiempo de convalecencia y las estancias hospitalarias.
Amplificación genómica:PCR convencional
La PCR (reacción en cadena de la polimerasa) se basa en la fabricación de nuevas cadenas de ADN simple a partir del original mediante enzimas específicas denominadas ADN polimerasas. Estas enzimas tienen la propiedad de realizar copias del ADN original a partir de una cadena simple de ADN siempre que tenga un iniciador (ADN de doble cadena). Los ciclos de desnaturalización y fabricación (replicación) consecutivos dan lugar a miles de copias del ADN original delimitado por los iniciadores (Saiki, et al., 1986). La PCR convencional se basa en amplificaciones mediante dos iniciadores, o cebadores, uno homólogo a la cadena 5′-3′ localizado al inicio del fragmento de ADN que se quiere amplificar y otro a la cadena 3′-5′ al final del fragmento.
Actualmente, existen infinidad de métodos de diagnóstico basados en PCR para la determinación de microorganismos en muestras humanas, en malaria el más conocido es el desarrollado por Georges Snounou (Snounou, 1996) conocido como el método de los cinco tubos. Estos métodos para patologías parasitarias como leishmaniasis, enfermedad del sueño, toxoplasmosis o criptosporidiasis han pasado del laboratorio de investigación a los hospitales y laboratorios de referencia basados generalmente en métodos de PCR anillada (nested PCR) para aumentar la sensibilidad del diagnóstico a partir de muestras clínicas (Cruz, et al., 2002). También se han incorporado o desarrollado métodos para la detección de marcadores de resistencia, virulencia y genotipificación molecular (Fuentes, et al., 2001).
Todos estos métodos tienen el inconveniente de ser individuales, es decir, nos dan una respuesta única, presencia o ausencia del patógeno buscado o del correspondiente gen que, en la mayoría de las ocasiones, no es suficiente, por lo que nuevos desarrollos de métodos basados en sistemas de detección múltiple los están sustituyendo.
Amplificación genómica: PCR múltiple
Las PCR múltiples son aquellas, en general, que en el proceso de amplificación participan más de dos iniciadores amplificando en un único tubo varias secuencias dianas, permitiendo la detección e identificación simultánea de distintos genes (Méndez-Álvarez y Pérez-Roth, 2004). En los últimos años estos métodos han proliferado en la literatura científica pero sus inicios son paralelos a la utilización de la PCR como herramienta para el diagnóstico a mediados de los años 90 del siglo pasado.
Podrían clasificarse en dos tipos:
i) aquellas que son PCR simples reunidas en un único proceso y en un único tubo y donde no existe competencia entre los iniciadores. En este grupo se incluirían PCR con control interno de reacción basado en otra pareja de iniciadores (Fuentes, et al., 1996). Estos se pueden hacer tan complejos como se desee, incluyendo numerosas parejas de iniciadores específicos para sus respectivas dianas, cuya mayor delimitación es la posibilidad de diferenciar los fragmentos obtenidos, generalmente por tamaño en geles de agarosa (Tenorio, et al., 1996), y
ii) aquellas que los iniciadores compiten entre ellos.
Semi-nested múltiple malaria PCR: un ejemplo La malaria es producida por protozoos parásitos del género Plasmodium. Cuatro especies afectan tradicionalmente a los humanos, P. falciparum, la forma más grave, P. vivax, P. ovale y P. malariae. Aunque son posibles las infecciones por otras especies (P. knowlesi).
En malaria es muy importante alcanzar un diagnóstico de especie correcto ya que la seriedad, patogenia y tratamiento de la enfermedad está estrechamente relacionada con la especie o especies de Plasmodium involucradas. El diagnóstico primario se hace por microscopía y, más recientemente, con el apoyo de pruebas de diagnóstico rápido. Ambos métodos tienen sus dificultades, además, adolecen de sensibilidad y la caracterización de infecciones mixtas es complicada.
El desarrollo de métodos moleculares intentaba suplir estas carencias. Se diseñaron diversos métodos, casi todos ellos basados en el gen ssurDNAl por dos razones; la sucesión de zonas muy conservadas y otras altamente variables, y la segunda que se disponía de un amplio número de secuencias en las bases de datos para diferentes especies de Plasmodium.
Dos estrategias se siguieron en el diseño, PCR individuales para cada Plasmodium humano (Snounou, 1996) o sistemas de detección múltiple (Rubio, et al., 1999).
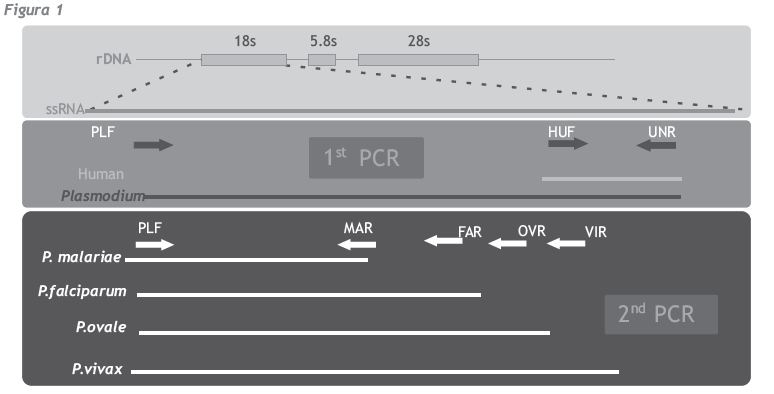
Este sistema consiste en dos amplificaciones múltiple encadenadas. La primera amplificación permite caracterizar Plasmodium sp. y, además, tiene un control interno de reacción. La segunda amplificación es una múltiple que permite identificar por tamaño del producto amplificado las cuatro especies de Plasmodium humanas (figura 1).
En la primera PCR, la pareja PLF-UNR amplifica el fragmento del gen en caso de que haya presencia de ADN de cualquier especie de Plasmodium, mientras que la pareja HUF-UNR amplifica un fragmento de 230 pares de bases correspondiente al fragmento del gen de humanos que se utiliza para comprobar que la reacción no se ha inhibido (control interno). En la segunda PCR, dependiendo de las especies involucradas en la infección, el producto de amplificación será de diferente tamaño: 250 pb para P. malariae, 399 pb para P. falciparum, 450 pb para P. ovale y 500 pb para P. vivax.
Las ventajas de este método de diagnóstico son su sensibilidad y su especificidad (Ataei, et al., 2011) pero, especialmente, la caracterización de las cuatro especies de Plasmodium que afectan a humanos en solo dos procesos de amplificación múltiple.
Diseño de una PCR múltiple: complejidad y dificultades
Diseñar PCR múltiple parece una tarea sencilla, en principio, la mayor dificultad parece únicamente el aumento del número de iniciadores que se deben diseñar. Pero la realidad es mucho más compleja. Entre otros factores es importante considerar los siguientes:
1. Se debe escoger un gen o regiones de genes con zonas variables y otras altamente conservadas del que se disponga de información en muchas o todas las especies de interés para nuestro objetivo.
2. Se deben localizar suficientes regiones del gen diana específicas para cada especie pero que, a su vez, el tamaño del fragmento teóricamente amplificado con las respectivas parejas de iniciadores se puedan distinguir con facilidad en geles de agarosa o el método de visualización escogido.
3. El diseño de los iniciadores debe cumplir las reglas mucho más estrictamente que para el diseño de PCR convencionales (Rubio, et al., 2002)
4. La temperatura de los iniciadores debe ser exactamente la misma para todos, en casos donde sea complejo la temperatura de alineamiento debe de ser compensada por la concentración de cada uno de ellos en la mezcla de amplificación.
Además, hay que considerar que pequeñas modificaciones de la concentración de sales en la reacción, incluyendo la concentración de Mg, pueden variar y descompensar los resultados, esto afecta directamente al uso cambio de enzimas y casas comerciales. La transferencia de este tipo de métodos entre laboratorios y su optimización es mucho más compleja que la trasferencia de métodos de PCR simples.
Otros sistemas múltiples en el diagnóstico parasitológico
Los sistemas múltiples no se circunscriben únicamente a sistemas de PCR convencional con múltiples iniciadores. El avance de los métodos moleculares ha permitido desarrollar métodos basados en nuevas tecnologías como son:
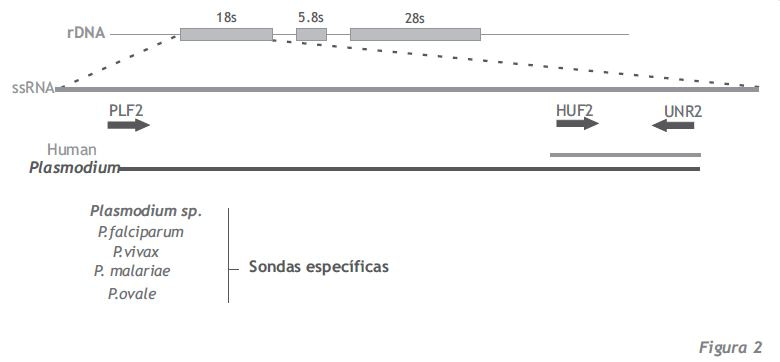
1. métodos de PCR a tiempo real, bien con sistemas de iniciadores múltiples o métodos mediante la detección de múltiples sondas (figura 2) (Ta, et al., 2010),
2. métodos de microarray o microchip mediante la fijación de múltiples sondas en soportes fijos (Palacios,et al., 2007), o
3. Métodos de pirosecuenciación de fragmentos previa PCR universal y fijación por múltiples sondas de los fragmentos amplificados (Sreekumar, et al., 2005).
Ventaja de los sistemas múltiples
La principal ventaja de estos sistemas es poder agrupar en un único proceso sistemas que antes requerían un número mayor de pasos, simplificando el proceso, ahorrando costes en tiempo, en personal y en recursos y lo que es más importante en diagnóstico, acortar el tiempo de respuesta.
Referencias Bibliográficas
1. Ataei S, Nateghpour M, Hajjaran H, Edrissian GH, Foroushani AR. High specificity of seminested multiplex PCR using dried blood spots on DNA Banking Card in comparison with frozen liquid blood for detection of Plasmodium falci-parum and Plasmodium vivax. J Clin Lab Anal. 2011;25:185-90.
2. Cruz I, Cañavate C, Rubio JM, Morales MA, Chicharro C, Laguna F, et al. Anested poly-merase chain reaction (Ln PCR) for diagnosing and monitoring Leishmania infantum infection in patients co-infected with human immunodeficiency virus. Trans R Soc Trop Med Hyg. 2002;96 (Suppl.1):S185-9.
3. Fuentes I, Rubio JM, Ramírez C, Alvar J. Genotypic characterization of Toxoplasma gondii strains associated with human toxoplasmosis in Spain: direct analysis from clinical samples. J Clin Microbiol. 2001;39:1566-70.
4. Fuentes I, Rodríguez M, Domingo CJ, del Castillo F, Juncosa T, Alvar J. Urine sample used for congenital toxoplasmosis diagnosis by PCR. J Clin Microbiol. 1996;34:68-71.
5. Méndez-Alvarez S, Pérez-Roth E. Multiplex PCR in clinical microbiology. Enferm Infecc Microbiol Clin. 2004;22:183-91.
6. Palacios G, Quan PL, Jabado OJ, Conlan S, Hirschberg DL, Liu Y et al. Panmicrobial oligonu-cleotide array for diagnosis of infectious disea-ses. Emerg Infect Dis. 2007;13:73-81.
7. Rubio JM, Benito A, Roche J, Berzosa PJ, García ML, Micó M, Edú M, Alvar J. Semi-nested, multiplex polymerase chain reaction for de-tection of human malaria parasites and eviden-ce of Plasmodium vivax infection in Equatorial Guinea. Am J Trop Med Hyg. 1999;60:183-7.
8. Rubio JM, Post RJ, van Leeuwen WM, Henry MC, Lindergard G, Hommel M. Alternative polymerase chain reaction method to identify Plasmodium species in human blood samples: the semi-nested multiplex malaria PCR (SnM-PCR). Trans R Soc Trop Med Hyg. 2002;96 (Suppl.1):S 199-204.
9. Saiki RK, Bugawan TL, Horn GT, Mullis KB, Erlich HA. Analysis of enzymatically amplified betaglobin and HLADQ alpha DNAwith allele-specific oligonucleotide probes. Nature. 1986;324:163-6.
10. Snounou G. Detection and identification of the four malaria parasite species infecting humans by PCR amplification. Methods Mol Biol. 1996;50:263-91.
11. Sreekumar C, Hill DE, Miska KB, Vianna MC, Yan L, Myers RL, Dubey JP. Genotyping and detection of multiple infections of Toxoplasma gondii using Pyrosequencing. Int J Parasitol. 2005;35:991-9.
12. Ta TT, Salas A, Ali-Tammam M, Martínez Mdel C, Lanza M, Arroyo E, Rubio JM. First case of detection of Plasmodium knowlesi in Spain by real time PCR in a traveller from Southeast Asia. Malar J. 2010;9:219.







