
Resistencia transferible a quinolonas en enterobacterias productoras de BLEE en un hospital pediátrico del Uruguay
Virginia García-Fulgueiras, Inés Bado, María Inés Mota, Luciana Robino, Nicolás F. Cordeiro, Adriana Varela, Gabriela Algorta, Gabriel Gutkind, Juan A. Ayala y Rafael Vignoli
1- Departamento de Bacteriología y Virología, Instituto de Higiene, Facultad de Medicina, Universidad de la República, Montevideo, Uruguay;
2- Laboratorio Central del Hospital Pereira Rossell-Ministerio de Salud Pública. Montevideo, Uruguay;
3- Cátedra de Microbiología, Facultad de Farmacia y Bioquímica, Buenos Aires, Argentina;
4- Centro de Biología Molecular “Severo Ochoa”, CSIC-UAM, Madrid, España
rvignoli@higiene.edu.uy
Introducción:
El control de las enfermedades infecciosas es un constante desafío, donde el tratamiento antimicrobiano constituye uno de los pilares fundamentales (1). En este sentido, las infecciones hospitalarias aumentan las tasas de morbimortalidad de los pacientes, el tiempo de hospitalización y elevan los costos asociados al cuidado de la salud (2,3).
La aparición de resistencia a los antimicrobianos en los microorganismos causantes de infecciones intrahospitalarias, es un problema creciente que amenaza con agotar los recursos terapéuticos disponibles. El conocimiento de los patrones de resistencia locales permite optimizar las terapias empíricas en base a los recursos disponibles.
Desafortunadamente, en Uruguay y otros países de la región, la información referente a la etiología y sensibilidad a antimicrobianos en las infecciones nosocomiales suele ser insuficiente. Dentro de los agentes responsables de infecciones nosocomiales se destacan los bacilos Gram negativos, en general multiresistentes, que causan infecciones severas tanto en población adulta como pediátrica (4). Entre estos, los más frecuentemente aislados son Acinetobacter baumannii (5), Pseudomonas aeruginosa (6), Klebsiella pneumoniae (7), Enterobacter cloacae (8), Proteus mirabilis y Escherichia coli (3).
La multirresistencia en estos microorganismos, puede deberse a la expresión de algún mecanismo de resistencia a múltiples drogas, como bombas de eflujo, (capaces de conferir resistencia a beta-lactámicos, fluoroquinolonas, macrólidos, sulfonamidas, tetraciclinas (excepto tigeciclina), trimetoprima, y cloranfenicol) (9) o a la adquisición de elementos reclutadores de genes de resistencia como transposones e integrones (10). Por otra parte, la diseminación de los microorganismos portadores y/o de los plásmidos de resistencia, es un fenómeno complejo y multifactorial que involucra, entre otros, la presión de selección que ejerce el consumo de antibacterianos, eventos de colonización cruzada o la presencia de reservorios ambientales de difícil erradicación (11,12).
El principal mecanismo de resistencia frente a las oxiiminocefalosporinas en enterobacterias es la producción de beta-lactamasas de espectro extendido (BLEEs). Este es un problema en constante aumento tanto en población adulta como pediátrica (13), frecuentemente asociado además a resistencia a aminoglucósidos y quinolonas (14,15).
La epidemiología de las BLEE en Latinoamérica es algo heterogénea, mientras que en Argentina enzimas como CTX-M-2 y PER-2 son las más frecuentes (16), en otros países de Sudamérica predominan otras variantes de CTX-M como CTXM-9 y CTX-M-15 (17).
En relación a los mecanismos de resistencia a quinolonas, podemos diferenciar entre cromosómicos o plasmídicos. Los primeros implican alteraciones en el sitio blanco o trastornos de permeabilidad (disminución en el número de porinas y/o sobreexpresión de bombas de eflujo). Las alteraciones del sitio blanco, involucran la acumulación de mutaciones en los genes que codifican para las girasas (gyrA y gyrB) y la topoisomerasa IV (parC y parE) (18).
Por otra parte, los mecanismos transferibles de resistencia a quinolonas (MTRQ), consisten hasta el momento en: a) enmascaramiento del sitio blanco; b) inactivación enzimática y c) eliminación activa.
El primero corresponde a genes denominados qnr (qnrA, qnrB, qnrS, qnrC y qnrD) (19-22) que codifican para proteínas pertenecientes a la familia de pentapéptidos repetidos. Estas proteínas impedirían, mediante unión a las girasas, la acción de las quinolonas bloqueando la formación del complejo ternario girasa-ADN-quinolona. La resistencia conferida por Qnr, puede cuadriplicar los niveles de concentración inhibitoria mínima de las quinolonas en relación a la cepa que no presenta dicha proteína (23).
Por otra parte la inactivación enzimática se produce por la acción de la enzima Aac(6´)Ib-cr, a través de la N-acetilación del anillo piperazilínico que presentan las fluoroquinolonas ciprofloxacina y norfloxacina. Esta enzima resulta de dos mutaciones (codones 102 y 179) en el gen aac(6′)-Ib responsable originalmente de resistencia a kanamicina, amikacina y tobramicina (24). De manera similar a Qnr, la presencia de esta enzima cuadriplica los niveles de resistencia a ciprofloxacina y norfloxacina, no afectando a otras quinolonas (fluoradas o no fluoradas). En Latinoamérica, la presencia de Aac(6´)Ib-cr ha sido reportada en distintos países (25,26,27), incluyendo Uruguay por nuestro grupo de trabajo (14).
Por último, dentro de las bombas de eflujo codificadas a nivel plasmídico, se encuentran QepA y OqxAB. La primera pertenece al grupo MSF (major facilitator subfamily) y presenta alta especificidad de sustrato por norfloxacina, ciprofloxacina y enrofloxacina. Hasta el momento, la incidencia reportada de QepA es baja (28) habiéndose descrito en Japón y Francia, y su expresión produce aumentos de hasta 40 veces los niveles de resistencia a los antibióticos antes mencionados (29). Por otro lado, OqxAB confiere resistencia a fluoroquinolonas entre otros compuestos, y pertenece a la familia RND (resistance nodulation cell-division) (30). La expresión de OqxAB en E.coli aumenta los niveles de concentración inhibitoria mínima (CIM) unas 8 veces al ácido nalidíxico y 16 veces a la ciprofloxacina (31).
Los genes que codifican para mecanismos transferibles de resistencia a quinolonas presentan una amplia distribución geográfica, principalmente dentro de la familia Enterobacteriaceae, y si bien la mayoría causan un bajo nivel de resistencia, favorecen y complementan la selección de mecanismos de resistencia adicionales.
Sin embargo, en los laboratorios clínicos, la interpretación de los resultados de sensibilidad suele hacerse sin la posibilidad de confirmación molecular.
Para ello en Sudamérica suelen tomarse como guías las elaboradas por el Clinical and Laboratory Standards Institute (CLSI) pudiéndose adoptar algunas modificaciones propuestas por la sub comisión de antimicrobianos de la Sociedad Argentina de Bacteriología Clínica (SADEBAC) (32).
Por su parte en Europa, desde 1999 se crea EUCAST (European Committee on Antimicrobial Susceptibility Testing) que define los criterios de interpretación para dicho continente.
En lo referente a enterobacterias, en los últimos años la aparición de resistencia transferible a quinolonas ha puesto en revisión los puntos de corte para dichos antibacterianos. Por otro lado en relación a la resistencia a oxiiminocefalosporinas, a partir del año 2010, tanto CLSI como EUCAST modifican sus pautas, recomendando interpretar la sensibilidad a cefalosporinas de tercera y cuarta generación de acuerdo a los resultados obtenidos, sin tener en cuenta la presencia o no de BLEE.
Sin embargo existen diferencias significativas entre una y otra pauta, así mientras CLSI define sensibilidad a ceftazidima y cefepima con valores de CIM 4mg/L y CIM 8mg/L respectivamente, EUCAST define un único valor de CIM 1mg/L para cefota-xima, cetriaxona, ceftazidima y cefepima.
Diferencias no tan drásticas pueden verse también, por ejemplo, en el caso de ciprofloxacina (Sensible CIM 0.5 mg/L EUCAST versus CIM 1mg/L CLSI) y amikacina (Sensible CIM 8 mg/L EUCAST versus CIM 16 mg/LCLSI).
Objetivo
El objetivo de este estudio fue determinar la prevalencia de MTRQ en enterobacterias productoras de BLEE en el hospital pediátrico del Uruguay y analizar los cambios de interpretación de sensibilidad según se utilicen las pautas CLSI y EUCAST elaboradas a partir de 2010.
Materiales y Métodos
Entre el 11 de mayo de 2009 al 10 de mayo de 2010, se recolectaron 651 aislamientos de enterobacterias en el Laboratorio del Hospital de Niños del Centro Hospitalario Pereira Rossell (CHPR). De éstas, 483 provenían del departamento de emergencia, 47 de la unidad de neonatología y 121 de pacientes de diferentes servicios del CHPR (salas pediátricas, unidad de cuidados intensivos, ortopedia, hematología/oncología, y departamento de cirugía). El 90.9% de estos aislamientos provenían de: urocultivos (76.2 %), hemocultivos (7.5%), heces (5.1%) y heridas quirúrgicas (2.1%).
Se incluyeron en el estudio un único aislamiento por paciente por episodio de hospitalización. En el caso de los pacientes re-hospitalizados, se estudiaron los nuevos aislamientos sólo si pertenecían a especies distintas o presentaban distintos perfiles de resistencia.
La identificación a nivel de especie y determinación de la susceptibilidad a antibacterianos fue realizado mediante el sistema automatizado VITEK® 2 Compact system (bioMérieux, Marcyl´Étoile, France).
La interpretación de los resultados se realizó de acuerdo a CLSI 201133 y EUCAST 2011 (http://www.eucast.org).
En el caso de enterobacterias productoras de BLEE, las concentraciones inhibitorias mínimas (CIM) a cefotaxima, amikacina y ciprofloxacina, fueron determinadas mediante E-test (AB bioMérieux, Suecia), de acuerdo a las recomendaciones del fabricante. La búsqueda y confirmación de BLEEs se realizó mediante disco difusión según recomendaciones de CLSI (33) independientemente del género y la especie, como se sugiere para áreas de alta prevalencia de enzimas CTX-M (16).
En aquellos aislamientos con resultados confirmatorios de BLEE, se realizó la búsqueda mediante PCR de la presencia de genes blaCTX-M, blaTEM, blaPER-2 y blaSHV utilizando primers específicos (34). Se repitió el ensayo para las muestras positivas utilizando la enzima Pfu ADN polimerasa (Fermentas, Life Sciences, Canadá) y se realizó la posterior secuenciación completa de ambas hebras.
Se buscó la presencia de los genes qnrA, qnrB, qnrS y aac(6′)Ib y la variante cr en las cepas productoras de BLEE mediante PCR y secuenciación de amplicones según lo descrito previamente (3).
Resultados
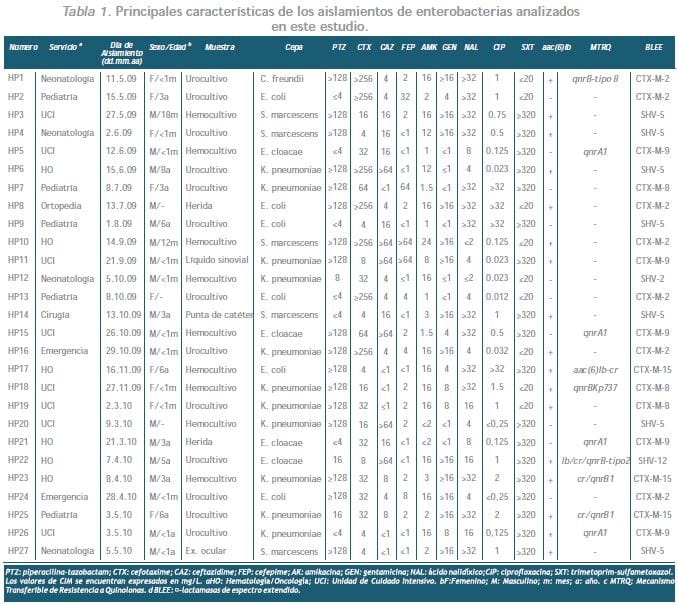
Del total de 651 aislamientos estudiados, 27 fueron caracterizados como productores de BLEE (21 provenientes de salas pediátricas, 4 de neonatología, y 2 del departamento de emergencia).
Un mismo paciente presentó dos aislamientos diferentes, en dos eventos distintos de hospitalización: en una primera instancia una cepa de E.coli productora de CTX-M-2 y en segundo lugar una cepa de K.pneumoniae productora de CTX-M-8.
La proporción de enterobacterias productoras de BLEE fue 17.3% (21/121), 8.5% (4/47) y 0.41% (2/483) para muestras obtenidas de salas pediátricas, unidad de neonatología y el servicio de emergencia, respectivamente.
Las enterobacterias productoras de BLEE fueron recuperadas a partir de muestras de orina (13), hemocultivos (9), herida quirúrgica (2), punta de catéter (1), líquido sinovial (1) y exudado ocular (1). Las distintas BLEE halladas en este trabajo se muestran en la Tabla 1.
El gen aac(6′)Ib se encontró en 17/27 aislamientos productores de BLEE, más específicamente blaCTXM- 15, blaCTX-M-2, blaCTX-M-8, blaCTX-M-9, blaSHV-5 ó blaSHV-12.
Cuatro aislamientos portaban la variante aac(6′) Ibcr, en tres casos junto a blaCTX-M-15 y en uno junto a blaSHV-12.
Dos aislamientos de K. pneumoniae, portadores de blaCTX-M-15/aac(6′)Ib-cr también presentaban qnrB mientras que la cepa de Enterobacter cloacae portadora de blaSHV-12/aac(6′)Ib-cr presentó también aac(6′)Ib y qnrB (ver tabla 1).
Adicionalmente un aislamiento de Serratia marcescens presentó la variante aac(6′)Ib7, mostrando una CIM a amikacina de solo 3 mg/L.
Nueve aislamientos presentaron variantes de qnr; a las tres mencionadas anteriormente se agregaron: tres aislamientos de Enterobacter cloacae y uno de Klebsiella pneumoniae portando qnrA1 ligado a blaCTX-M-9, una cepa de Citrobacter freundii con qnrB tipo 8 y blaCTX-M-2, y una cepa de Klebsiellapneumoniae portando blaCTX-M-8 y una nueva variante de qnrB. En relación a este último aislamiento, los transconjugantes obtenidos en ensayos de conjugación (TcKp737) mostraron un aumento de aproximadamente 12 veces en la CIM a ciprofloxacina (0.38 mg/L vs 0.032 mg/L de la cepa receptora E.coli J53-2).
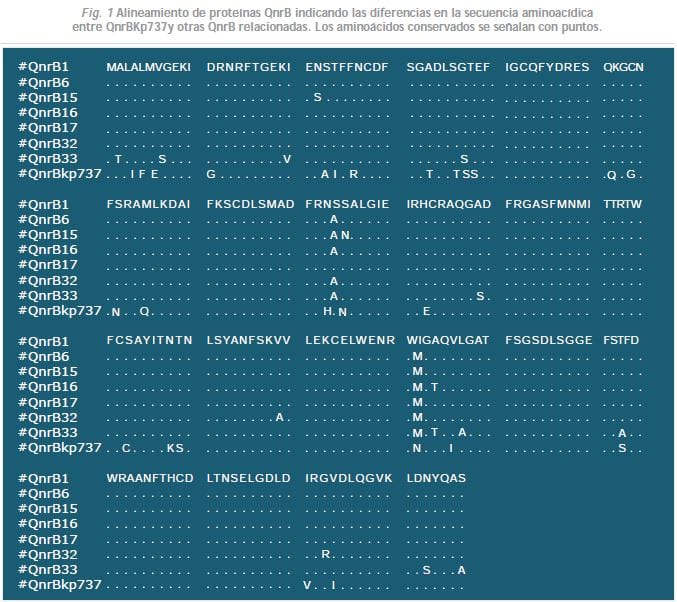
La secuencia nucleotídica parcial de la nueva variante de qnrB (606pb), obtenida con los primers qnrBR34 y tnpAISEcp135, mostró un 77% de similitud con qnrB17, mientras que la secuencia aminoacídica deducida mostró 87% de identidad con l proteína correspondiente, presentando 26 diferencias con QnrB1, 21 de las cuales no están descritas en el sitio web http://www.lahey.org/qnrStudies (ver fig.1).
Análisis de recomendaciones CLSI/EUCAST
De acuerdo a los criterios utilizados para la detección de BLEE y a la confirmación molecular realizada, detectamos 27 aislamientos de enterobacterias productoras de BLEE.
Siguiendo las recomendaciones EUCAST 2011, de las 27 cepas con BLEE confirmadas, 5 hubiesen sido informadas como sensibles a ceftazidima (18.5%) y 11 como sensibles a cefepima (40.7%).
Con respecto a cefotaxima, todas las cepas con BLEE hubiesen sido informadas como resistentes a dicho antibacteriano.
El mismo análisis realizado de acuerdo a las normas CLSI arroja que 12 de 27 cepas (44.4%) debieron informarse como sensibles a ceftazidima y 23 de 27 (85.2%) sensibles a cefepima, siendo las 27 resistentes a cefotaxima. Globalmente, en 25/27 (92.6%) de los casos las cepas resultarían ser susceptibles a cefepima y/o ceftazidima (ver tabla 1).
En relación a ciprofloxacina, 7/27 cepas (25.9%) fueron catalogadas como sensibles de acuerdo a CLSI, mientras que según las normas EUCAST las mismas serían resistentes o intermedias. En dos de tales cepas se detectó algún mecanismo transferible de resistencia a quinolonas. Adicionalmente, en cuatro aislamientos (todos portadores de blaCTX-M-9 y qnrA1) los valores de CIM estaban por debajo de ambos puntos de corte y en tres casos incluso por debajo del punto de determinación del sistema VITEK, presentando una CIM a ciprofloxacina de 0.125 mg/L.
Algo similar ocurre con la sensibilidad a amikacina donde 13/27 aislamientos (48.2%) interpretados como sensibles de acuerdo a CLSI, presentarían sensibilidad intermedia según EUCAST. En 11 de estos aislamientos se detectó la presencia de alguna variante alélica de aac(6′)-Ib.
Conclusiones y discusión
La epidemiología de las BLEE en Uruguay se encuentra en constante cambio, de acuerdo a datos previos de nuestro país (34). En este sentido, TX-M-2 representa solo un 26% (7/27) de las enzimas detectadas en nuestro trabajo. De las 20 restantes, 11 fueron CTX-M de grupos diferentes a CTX-M-2 (3 CTX-M-8, 5 CTX-M-9 y 3 CTX-M-15) y 9 fueron variantes de SHV (7 SHV-5, 1 SHV-2 y 1 SHV-12).
Conjuntamente con estas BLEE se produjo también un aumento en la circulación de resistencia transferible a quinolonas mediada por distintos mecanismos, e incluso por acumulación de los mismos. Las cepas productoras de QnrB y Aac(6′)Ib-cr son capaces de transferir plásmidos que confieren en un solo paso valores de CIM a CIP de 1 mg/L a la cepa receptora (datos no mostrados).
Es llamativa la tendencia ascendente de asociación entre BLEE y MTQR en los últimos meses de nuestro estudio. Si comparamos la relación MTQR/BLEE entre el primero el segundo semestre (periodo mayo-octubre 2009 contra noviembre-abril 2010), se observa que en el primer período la relación era aproximadamente 1/5 (3 MTQR/16 BLEE) contra una relación 1/1 (10 MTQR/11 BLEE) en el segundo período. Este aumento, evidenciable desde abril de 2010 en que comienzan a aislarse cepas que presentan más de un mecanismo de resistencia a quinolonas, estaría favorecido por un fenómeno de “capitalismo genético o microbiológico” en el cual aquellas cepas que ya presentan algún mecanismo de resistencia tienen mayor tendencia a seguir acumulando más determinantes de resistencia.
De las 27 cepas portadoras de BLEE estudiadas, en 14 existiría una opción terapéutica dentro de las oxiiminocefalosporinas de acuerdo a las pautas EUCAST, mientras que dicho número asciende a 25 de acuerdo a CLSI.
En tal sentido, los microbiólogos latinoamericanos deberían plantearse la discusión de pautas de interpretación de sensibilidad autóctonas, adaptadas los perfiles de susceptibilidad presentes en nuestra región. Esto es particularmente importante para disminuir el uso de carbapenemes en una región azotada por la presencia de KPC-2 (36,37).
Paralelamente, médicos y microbiólogos deberían definir en conjunto la posibilidad de identificar pacientes que puedan comenzar a ser tratados con oxiiminocefalosporinas aún en presencia de BLEE, de acuerdo a las nuevas recomendaciones internacionales. Por ejemplo, en nuestro estudio los 13 aislamientos obtenidos de infección urinaria eran sensibles a alguna oxiiminocefalosporina.
El conocimiento de las propiedades farmacocinéticas/farmacodinámicas (pK/pD) de los antibacterianos ha modificado significativamente los puntos de corte y la forma de dosificar un conjunto de antimicrobianos. Sería importante acumular experiencia a nivel regional sobre el funcionamiento clínico de estas nuevas recomendaciones
Financiación
Este trabajo fue financiado parcialmente mediante fondos de la Comisión Sectorial de Investigación Científica (CSIC Uruguay) para R.V., así como con fondos FP7-HEALTH-2007-223431 de European Community and BFU2009-09200 del Ministerio de Ciencia e Innovación/Programa BioFundamental, España, para J.A.A.
G.G. es Miembro de la Carrera del Investigador Científico (CONICET, Argentina).






