Inmunofluorescencia indirecta versus reacción de polimerasa en cadena para el diagnóstico de virus respiratorios en niños ingresados en un hospital de la Región Metropolitana
Pablo Corvalán L.1 Guisselle Arias B.2 Paola Morales S.3 Raquel González M.4 Jaime Inostroza S.5 Loreto Fuenzalida I.2
1Estudiante de Enfermería, Facultad de Ciencias de la Salud, Universidad Autónoma de Chile. Santiago, Chile.
2Instituto de Ciencias Biomédicas, Facultad de Ciencias de la Salud, Universidad Autónoma de Chile. Santiago, Chile.
3Centro Salud Integral, Universidad Autónoma de Chile. Santiago, Chile.
4Laboratorio de Microbiología, Hospital Dr. Exequiel González Cortés. Santiago, Chile.
5Centro Jeffrey Modell para Diagnóstico e Investigación en Inmunodeficiencias Primarias, Centro de Excelencia en Medicina Traslacional,Facultad de Medicina, Universidad de La Frontera, Temuco.
Revista chilena de infectología – versión impresa ISSN 0716-1018 – Rev. chil. infectol. vol.36 no.1 Santiago feb. 2019
http://dx.doi.org/10.4067/S0716-10182019000100026 – LABORATORIO E INFECTOLOGÍA
Correspondencia: Loreto Fuenzalida Inostroza lfuenzalidai@uautonoma.cl
Bernanda Morín #488, 2° Piso, Providencia, Santiago – Chile – Fono/Fax: (56-2) 23413539 – revinf@sochinf.cl
Resumen
Introducción: La temprana detección viral en infecciones respiratorias agudas (IRA) es esencial para establecer una terapia apropiada y prevenir el contagio intrahospitalario.
Objetivo: Comparar la eficacia de la técnica de inmunofluorescencia indirecta (IFI) con la reacción de polimerasa en cadena (RPC) para identificar virus respiratorios en niños hospitalizados por IRA.
Métodos: Se incluyeron 47 aspirados nasofaríngeos de niños ≤ 2 años con IRA. La IFI incluyó virus respiratorio sincicial (VRS), adenovirus, influenza A y B y parainfluenza. La RPC incluyó, además, la detección de metapneumovirus, enterovirus/rinovirus, bocavirus y coronavirus. Se estimó sensibilidad, especificidad, valor predictor positivo y negativo (VPP/VPN) y correlación kappa para VRS mediante IFI en comparación a la RPC.
Resultados: La IFI detectó únicamente VRS (29; 61,7%). La RPC detectó diversos virus, entre ellos VRS en 26 casos (55,3%), seguido por bocavirus (29,8%), enterovirus/ rinovirus (21,3%), adenovirus (14,9%) y parainfluenza (4,3%) entre otros, con 35,5% de co-infección. La IFI presentó sensibilidad: 85,7%, especificidad: 73,6%, VPP: 82,7%, VPN: 77,7% y kappa: 0,5990 (IC 95%; 0,36360,8346) para VRS.
Conclusión: La IFI presenta buena sensibilidad, pero moderada especificidad para VRS. Sin embargo, falla en la detección de otros virus respiratorios. La introducción de RPC permitiría mejorar el diagnóstico etiológico de las IRA de origen viral.
Palabras clave: Virus respiratorios, inmunofluorescencia indirecta, reacción de polimerasa en cadena, infección respiratoria aguda
Abstract
Background: Early viral detection in acute respiratory infections (ARI) is essential to establish appropriate therapy and prevent nosocomial transmission.
Objective: To compare the efficacy of indirect immunofluorescence technique (IIF) with the polymerase chain reaction (PCR) to identify respiratory viruses in children hospitalized for ARI.
Methods: 47 nasopharyngeal aspirates of children ≤ 2 years with ARI were included. IFI included respiratory syncytial virus (RSV), adenovirus, influenza A and B and parainfluenza. PCR also included the detection of metapneumovirus, enterovirus/rhinovirus, bocavirus and coronavirus. Sensitivity, specificity, positive and negative predictive value (VPP/NPV) and kappa correlation for RSV were estimated by IIF compared to PCR.
Results: The IIF detected only RSV (29; 61.7%). PCR detected several viruses, including RSV in 26 cases (55.3%), followed by bocavirus (29.8%), rhinovirus/enterovirus (21.3%), adenovirus (14.9%) and parainfluenza (4,3%) among others, with 35.5% of coinfection. The IIF presented sensitivity: 85.7%, specificity: 73.6%, PPV: 82.7%, NPV: 77.7% and kappa: 0.5990 (95% CI, 0.3636-0.8346) for RSV.
Conclusion: The IIF presents good sensitivity, but moderate specificity for RSV. However, IIF fails to detect other respiratory viruses. The introduction of PCR would improve the etiological diagnosis of ARI of viral origin.
Keywords: Respiratory viruses; indirect immunofluorescence; polymerase chain reaction; acute respiratory infection
Introducción
Las infecciones respiratorias agudas (IRAs) representan una importante causa de morbimortalidad en pediatría1,2. Según la Organización Mundial de la Salud (OMS), la incidencia de las IRA ha ido en aumento en el mundo; en el año 2008 la incidencia anual fue de 156 millones de casos, siendo 43% lactantes o niños pequeños1; en el año 2011, la estimación de la incidencia según la OMS fue de 450 millones de casos de IRA, que se asociaron con aproximadamente 4 millones de muertes, representando 7% de todas las muertes relacionadas con enfermedades3.
Al año, los niños pequeños y lactantes, pueden presentar entre seis y ocho exacerbaciones respiratorias por infecciones, siendo los virus la etiología más frecuente4,5. Estas infecciones representan una gran parte de todas las visitas a centros de salud pediátricos y es una gran carga económica para la sociedad6. Dentro de los virus causantes de IRA destacan el virus respiratorio sincicial (VRS), virus influenza A y B (IA e IB), virus parainfluenza (VPI), adenovirus (ADV) y rinovirus (RV)7,8. Además, se han descrito nuevos virus respiratorios humanos en los últimos años, incluidos metapneumovirus (HMPV)9, bocavirus (HBOV)10 y coronavirus (HCOV)11.
El diagnóstico de las infecciones respiratorias virales se basa en el uso de métodos convencionales como el aislamiento por cultivo celular y la detección antigénica mediante inmunofluorescencia (IF). Aunque estos métodos son efectivos y a menudo complementarios, tienen algunas desventajas. La IF se encuentra limitada a ocho virus o menos (ADV, IA, IB, VPI 1-3, VRS, HMPV) y puede carecer de sensibilidad según el título viral, la edad del paciente y el tiempo de prueba en relación con el inicio de los síntomas12. Sin embargo, es ampliamente usada en hospitales regionales y laboratorios de referencia debido a su bajo costo y su capacidad para procesar un alto número de muestras, simultáneamente, en un corto período de tiempo13. El aislamiento o cultivo viral, si bien ha sido denominado el “estándar de oro”, es una técnica costosa y un número significativo de muestras en pacientes clínicamente compatibles con infección respiratoria viral resultan negativos14.
Por otra parte, la reacción de polimerasa en cadena (RPC) ha demostrado ser altamente sensible y específica en comparación con los métodos convencionales como la detección de antígenos y/o el cultivo viral. El aumento adicional de los rendimientos se debe a la detección de virus respiratorios que no se detectan eficazmente por métodos convencionales (p. ej.: ADV), a los que se agrega en los test que utilizan RPC, la pesquisa de RV y HCOV. En niños con IRA, las tasas de positividad viral varían de 40 a 45% por IF a 67-88% por métodos moleculares, por lo que, actualmente, se considera la RPC como la técnica de referencia15.
Este estudio tiene como objetivo comparar la eficacia de una técnica de inmunofluorescencia indirecta (IFI) con una técnica de detección molecular para la identificación virus respiratorios en niños hospitalizados por IRA.
Materiales y Métodos
Diseño general
Estudio prospectivo, descriptivo, transversal, cuyo objetivo es comparar dos técnicas, una inmunofluorescencia y una RPC, para la detección de virus respiratorios. Se incluyeron niños bajo 2 años de edad, con infección respiratoria aguda baja (IRAB) ingresados en el Hospital Dr. Exequiel González Cortés (Santiago, Chile), entre los meses de junio y julio de 2017. Se recolectó una muestra de aspirado nasofaríngeo, además de datos clínicos y demográficos de cada paciente: edad, sexo, vacunas, lactancia materna, días de hospitalización, días de oxigenoterapia, ingreso a Unidad de Cuidados Intensivos (UCI) o Unidad de Tratamientos Intensivos (UTI).
Muestras respiratorias
Se recogió, aproximadamente, 3 mL de aspirado nasofaríngeo siempre dentro de las primeras 24 h de ingreso. Se aspiraron secreciones de ambas fosas nasales y fueron depositadas en 3 mL de solución salina. La muestra fue homogenizada y dividida en alícuotas, almacenándose a −80 °C hasta su determinación viral.
Extracción de ácidos nucleicos
Se utilizaron 150 µL de aspirado nasofaríngeo para la extracción de ADN y ARN viral mediante columna NucleoSpin® RNA Virus (MACHEREY-NAGELGmbH & Co. KG., Düren, Alemania). Se agregó proteínasa K para extraer ADN, de acuerdo con lo recomendado por el fabricante.
Detección de virus respiratorios
La técnica de IFI incluyó los siguientes virus: VRS, ADV, VPI e IA y B (Diagnostics Hybrids, Athens, OH, EE. UU.) entregado por el Instituto de Salud Pública de Chile (ISP) a los centros de referencia. Las muestras fueron procesadas según las indicaciones del ISP. La técnica de RPC se realizó con el kit comercial Argene Real-Time PCR (bioMérieux, Francia) que incluyó: VRS, HMPV, HCOV, VPI, IA, IB, enterovirus/rinovirus (EV/RV), HBOV y ADV, según instrucciones del fabricante. Brevemente, 10 µL del eluído de cada muestra fue mezclado con 15 μL de premix de amplificación de cada virus y 0,15 μL de RT. La amplificación de los diferentes genes virales y la detección de fluorescencia se realizaron utilizando el instrumento LightCycler® 96 (Roche Diagnostics, Switzerland) con el siguiente programa: retro-transcripción 1 ciclo a 50 °C/5 min, activación 1 ciclo a 95 °/15 min y 45 ciclos de 95 °C/10 seg, 60 °C/40 seg, 72 °C/25 seg.
Además, los resultados discordantes entre ambas técnicas fueron analizadas mediante una segunda RPC con la cual fue posible, además de tener una confirmación del resultado, cuantificar la carga viral en caso de que la muestra presentase VRS, considerando una elevada carga viral valores sobre 2,5 × 105 copias/mL16.
El ARN sintético se obtuvo por transcripción in vitro (RevertAid H Minus First, Thermo Scientific®, USA) después de la clonación del gen N en el vector pGEM-T-Easy (PROMEGA), a partir de una muestra positiva para VRS y transformado en Escherichia coli top ten. Con posterioridad a la purificación, se realizaron diluciones de ARN como patrones cuantitativos durante la reacción de RPC cuantitativa. La muestra se llevó a una concentración conocida de 100 ng/μL. Posteriormente se realizó una RPC con el kit Go taq® Green Master Mix (Promega, USA) con el siguiente protocolo de amplificación: 94 °C/5 min, 94 °C/20 seg, 58 °C/20 seg, 72 °C/20 seg y 72 °C/7 min y 35 ciclos.
La RPC cuantitativa se realizó utilizando el kit Takyon™ Rox Probe MasterMix dTTP blue (Eurogentec, USA), utilizando los partidores con sonda N1 (5’-GGAACAAGTTGTTGAGGTTTATGAATATGC3’) y N2 (CTTCTGCTGTCAAGTCTAGTACACTGTAGT-3’)17. Las condiciones de la reacción de RPC fueron: activación 95 °C/3 min; 40 ciclos, denaturación 95 °C/10 seg; anillado y extensión 60 °C/30 seg. Para conocer la cantidad efectiva de copias o moléculas de ADN viral contenidas en cada una de las muestras fue necesario contar con un gen endógeno ARNasa P, utilizando el kit TaqMan®RNase P (Applied Biosystems, UK). Se interpolaron los valores de Cq y se normalizaron según la siguiente fórmula: Cq muestra normalizada = (Cq muestra viral) × (Cq RNasa P de la muestra)/valor promedio de Cq de todas las muestras de RNasaP)18.
Análisis estadístico
Se determinó sensibilidad, especificidad, valor predictor positivo (VPP), valor predictor negativo (VPN) e índice kappa mediante el programa VassarSTats. Se consideró un valor de p < 0,05 como diferencia estadísticamente significativa, con un intervalo de confianza (IC) de 95%.
La investigación fue aprobada por el Servicio de Salud Metropolitano Sur (SSMS) y por el Comité Ético Científico de la Universidad Autónoma de Chile. Todos los padres o tutores de los niños firmaron un consentimiento informado de participación en el estudio.
Resultados
Se analizaron 47 muestras de aspirado nasofaríngeo provenientes de 47 pacientes ingresados por patología respiratoria aguda. En la Tabla 1 se muestran datos sociodemográficos y clínicos de los pacientes.
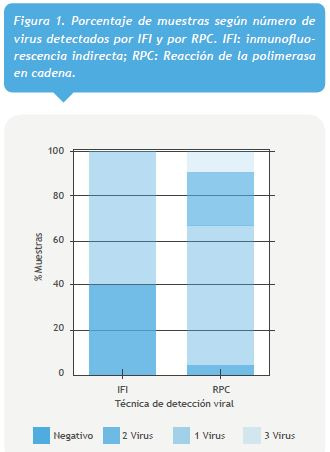
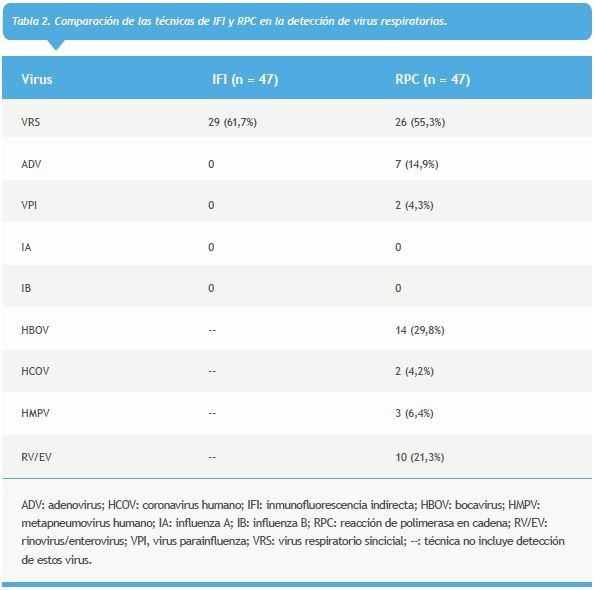
Del total de muestras analizadas mediante IFI, 29 (61,7%) fueron positivas para VRS como único agente etiológico y las restantes 18 (38,3%) muestras fueron negativas para virus respiratorios (Figura 1). Por el contrario, mediante RPC se encontraron 45 muestras positivas para la presencia de al menos un virus respiratorio (95,7%) y sólo dos muestras fueron negativas (4,2%). De las 45 muestras positivas por RPC, 29 presentaron sólo un virus (61,7%), 11 muestras presentaron dos virus (23,4%) y cinco muestras presentaron tres virus (10,6%). Entre los principales virus detectados por RPC se encuentra VRS en 26 casos (55,3%), HBOV en 14 (29,8%) y RV/EV en 10 (21,3%), tal como se detalla en la Tabla 2. La técnica de IFI incluye la detección de ADV y VPI; sin embargo, no fue capaz de detectar ninguno de los siete casos positivos por RPC para ADV (14,9%) ni los dos para VPI.
Se realizó únicamente el análisis de la eficiencia de la técnica de IFI para la detección de VRS, debido a que es el único virus que se detecta por esta técnica, usando como método de referencia la RPC (Tabla 3). Como se puede apreciar, mediante IFI, cinco de los 47 casos fueron falsos positivos, mientras que cuatro fueron falsos negativos, los que fueron corroborados por RPC con transcriptasa reversa (RPC-TR) cuantitativa. Las cinco muestras falsamente positivas para VRS, mediante IFI, fueron negativas mediante RPC-TR. Por otra parte, las cuatro muestras que fueron falsas negativas por IFI, fueron positivas por la nueva técnica, presentando una mediana de 5,0 × 105 copias/mL (rango: 5,0 × 104 − 2,5 × 108 copias/mL).
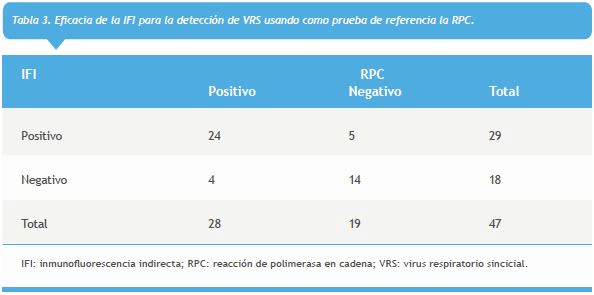
Los indicadores de calidad de la IFI para la detección de VRS mostraron un valor aceptable para sensibilidad (85,7%) y un menor valor para especificidad (73,6%). La concordancia obtenida entre la IFI y RPC fue moderada19, con un índice de kappa (k) de 0,5990 (IC 95%; 0,36360,8346) (Tabla 4).

Discusión
Las infecciones respiratorias virales afectan principalmente a niños bajo 5 años de edad, se propagan fácilmente en las salas de pediatría y causan infecciones asociadas a la atención en salud (IAAS)20.
La detección precisa del agente causal es importante para orientar los cuidados, prevenir su propagación nosocomial, disminuir los costos hospitalarios y la duración de la estadía hospitalaria21, como también proporcionar vigilancia no tan sólo en pacientes puntuales, sino que también como una herramienta para la vigilancia epidemiológica de virus respiratorios.
En el presente estudio se incluyeron 47 niños bajo 2 años de edad, hospitalizados por IRA. Mediante la prueba de IFI se encontraron 29 pacientes positivos para VRS, siendo aislados en sala de cohorte para VRS. De estos 29 pacientes, la técnica de RPC identificó que 24 pacientes eran positivos para VRS y los cinco pacientes restantes eran negativos para la presencia de dicho virus. Los 18 pacientes que fueron negativos para VRS mediante IFI, fueron derivados a sala común. En este grupo, la técnica de RPC, identificó que sólo 14 pacientes eran verdaderamente negativos por lo que cuatro pacientes infectados con VRS fueron dejados en sala común. Por otra parte, la carga viral de los pacientes con resultado negativo por IFI para VRS es alta16, por lo que la falla de la IFI no se debe a una baja carga viral, sino que a una insuficiente sensibilidad de la técnica. La mala gestión del aislamiento en cohorte por VRS al disponer de una técnica de sensibilidad limitada hace que los pacientes en aislamiento en cohorte o en sala común puedan potencialmente infectarse con VRS.
Por otra parte, se observó que la técnica de IFI no fue capaz de detectar ADV a pesar de que lo incluía en su panel de detección. Este virus fue detectado en siete de los 47 menores (14,9%) estudiados mediante RPC. A pesar de que la IFI tiene baja sensibilidad para la detección de ADV, es posible que no se hayan detectado casos positivos debido al bajo número de muestras estudiadas. La diseminación de la infección por ADV es alta y prolongada en el tiempo y se justifica extender las medidas preventivas de IAAS, en especial en recintos cerrados y de largas estadías de sus pacientes, especialmente en lactantes20, destacándose el hecho que, si el paciente tiene dos años o menos, presenta un mayor riesgo de contraer IAAS por este virus. Los brotes que involucran a poblaciones o personas con enfermedad respiratoria previa pueden ser particularmente graves, con altas tasas de morbimortalidad22, ya que compromete otros órganos23,24 comprobando la necesidad del aislamiento clínico.
Llamativamente, no se detectaron muestras positivas de IA e IB, por ninguna de las dos técnicas. Esto puede ser debido al bajo número de muestras analizadas, pero también a que todos los niños estudiados se encontraban con las vacunas al día según el calendario chileno, incluyendo la vacuna contra virus influenza A y B.
La técnica de RPC fue también capaz de detectar diferentes agentes virales, encontrándose co-infección viral en 35,5% de las muestras. Estos resultados concuerdan con lo descrito por Martin y cols., quienes encontraron que ADV, RV, PIV y HBOV son detectados con mayor frecuencia en co-infección que otros virus. Las co-infecciones están relacionadas con el período prolongado de permanencia que tiene el virus en la mucosa del tracto respiratorio25. Broccolo y cols., también apoyan la hipótesis de que la permanencia del virus juega un papel en la alta frecuencia de co-infecciones con otros patógenos26. El estudio de Harada y cols., indica que la co-infección con VRS y otros virus respiratorios como ADV, RV, VPI, HBOV y RV/EV entre otros, no es rara y que las co-infecciones pueden aumentar la gravedad de la enfermedad por VRS en niños27. Cabe destacar que una alta carga de virus de HBOV, podría conducir a un síntoma más grave del tracto respiratorio inferior28 mientras que EV es asociado con una alta morbilidad en niños con asma y co-morbilidades29.
En conclusión, la IFI tiene una buena sensibilidad, pero insuficiente comparada con la RPC para la detección de VRS; además, falla en la detección de otros virus respiratorios. Si bien la RPC es una técnica más costosa que las técnicas convencionales, permitiría un aumento en la detección de agentes etiológicos, con la posibilidad de tomar medidas preventivas para evitar el desarrollo de infecciones intrahospitalarias. Por esto, sería pertinente actualizar las técnicas de diagnóstico etiológico viral en hospitales públicos de nuestro país.
Estudio financiado por CONICYT Fondecyt 11150599 y Beca Iniciación Científica, Vicerrectoría de Investigación y Postgrado, Universidad Autónoma de Chile.
Agradecimientos. Al personal del Hospital Dr. Exequiel González Cortés, pacientes y padres que participaron en el estudio. CONICYT Proyecto Fondecyt N°11150599. Marjorie Reyes por ayuda en formato de figuras y bibliografía. Beca Iniciación Científica, Vicerrectoría de Investigación y Postgrado, Universidad Autónoma de Chile.
Referencias Bibliográficas
1.- Rudan I, Boschi-Pinto C, Biloglav Z, Mulholland K and Campbell H. Epidemiology and etiology of childhood pneumonia. Bull World Health Org 2008; 86 (5): 408-16. PMCID: PMC2647437. [ Links ]
2.- Liu L, Oza S, Hogan D, Perin J, Rudan I, Lawn J E, et al. Global, regional, and national causes of child mortality in 2000-13, with projections to inform post-2015 priorities: an updated systematic analysis. Lancet 2015; 385 (9966): 430-40. DOI: 10.1016/S0140-6736(14)61698-6. [ Links ]
3.- Ruuskanen O, Lahti E, Jennings LC, Murdoch D R. Viral pneumonia. Lancet 2011; 377 (9773): 1264-75. DOI: 10.1016/S0140-6736(10)61459-6. [ Links ]
4.- Mäkela M J, Puhakka T, Ruuskanen O, Leinonen M, Saikku P, Kimpimaki M, et al. Viruses and bacteria in the etiology of the common cold. J Clin Microbiol 1998; 36 (2): 539-42. PMCID: PMC104573. [ Links ]
5.- García-García M L, Ordobas G M, Calvo R C, González A M, Aguilar R J, Arregui S A. Infecciones virales de vías aéreas inferiores en lactantes hospitalizados: etiología, características clínicas y factores de riesgo. An Esp Pediatr 2001; 55: 1017. http://www.analesdepediatria.org/es-pdf-S1695403301776448. [ Links ]
6.- Fendrick A M, Monto A S, Nightengale B, Sarnes M. The economic burden of noninfluenza-related viral respiratory tract infection in the United States. Arch Intern Med 2003; 163 (4): 487-94. PMID: 12588210. [ Links ]
7.- Kusel M M, de Klerk N H, Holt P G, Kebadze T, Johnston S L, Sly P D. Role of respiratory viruses in acute upper and lower respiratory tract illness in the first year of life: a birth cohort study. Pediatr Infect Dis J 2006; 25: 6806. DOI: 10.1097/01.inf.0000226912.88900.a3. [ Links ]
8.- Brittain-Long R, Nord S, Olofsson S, Westin J, Anderson LM and Lindh M. Multiplex real-time PCR for detection of respiratory tract infections. J Clin Virol 2008; 41: 53-6. DOI:10.1016/j.jcv.2007.10.029. [ Links ]
9.- Fuenzalida L, Fabrega J, Blanco S, Del Mar Martínez M, Prat C, Pérez M, et al. Usefulness of two new methods for diagnosing metapneumovirus infections in children. Clin Microbiol Infect 2010; 16 (11): 1663-8. DOI: 10.1111/j.1469-0691.2010.03192.x. [ Links ]
10.- Allander T, Tammi M T, Eriksson M, Bjerkner A, Tiveljung-Lindell A, Andersson B. Cloning of a human parvovirus by molecular screening of respiratory tract samples. Proc Natl Acad Sci USA 2005; 102 (36): 12891-6. DOI: 10.1073/pnas.0504666102. [ Links ]
11.- Coleman C M, Frieman M B. Coronaviruses: important emerging human pathogens. J Virol. 2014; 88 (10): 5209-12. DOI: 10.1128/JVI.03488-13. [ Links ]
12.- Chartrand C, Leeflang M M G, Minion J, Brewer T, Pai M. Accuracy of rapid influenza diagnostic tests: a meta-analysis, Ann Intern Med 2012; 156: 500-11. DOI: 10.7326/0003-4819-156-7-201204030-00403. [ Links ]
13.- Wilhelm J, Yubero J, Fuentes, C, Ríos, P, Leyton O, Reyes F. Evaluation of three laboratory methods diagnostic sensitivity in influenza A infection: RIDT, DFA and DFA with cytocentrifugation versus RT-PCR. Rev Chilena Infectol 2014; 31 (6): 690-3. DOI: 10.4067/S0716-10182014000600009. [ Links ]
14.- Gharabaghi F, Tellier R, Cheung R Collins C, Broukhanski G, Drews SJ, et al. Comparison of a commercial qualitative real-time RT-PCR kit with direct immunofluorescence assay (DFA) and cell culture for detection of influenza A and B in children. J Clin Virol 2008; 42: 190-3. DOI: 10.1016/j.jcv.2008.01.013. [ Links ]
15.- Fairchok M P, Martin E T, Chambers S, Kuypers J, Behrens M, Braun L E, et al. Epidemiology of viral respiratory tract infections in a prospective cohort of infants and toddlers attending daycare. J Clin Virol 2010; 49: 16-20. DOI: 10.1016/j.jcv.2010.06.013. [ Links ]
16.- Campanini G, Percivalle E, Baldanti F, Rovida F, Bertaina A, Marchi A, et al. Human respiratory syncytial virus (hRSV) RNA quantification in nasopharyngeal secretions identifies the hRSV etiologic role in acute respiratory tract infections of hospitalized infants. J Clin Virol 2007; 39 (2):119-24. DOI: 10.1016/j.jcv.2007.03.009. [ Links ]
17.- Luchsinger V, Ampuero S, Palomino M A, Chnaiderman J, Levican J, Gaggero A, et al. Comparison of virological profiles of respiratory syncytial virus and rhinovirus in acute lower tract respiratory infections in very young Chilean infants, according to their clinical outcome. J Clin Virol 2014; 61 (1): 138-44. DOI: 10.1016/j.jcv.2014.06.004. [ Links ]
18.- Duchamp M B, Casalegno J S, Gillet Y, Frobert E, Bernard E, Escuret V, et al. Pandemic A(H1N1) 2009 influenza virus detection by real time RT-PCR: is viral quantification useful? Clin Microbiol Infect 2010; 16 (4): 317-21. DOI: 10.1111/j.1469-0691.2010.03169.x. [ Links ]
19.- Altman D G. Practical Statistics for Medical Research. 1 ed. Chapman & Hall/CRC Texts in Statistical Science 1991. [ Links ]
20.- Flores C, Méndez M, Astudillo C, Cerda H, Espinoza T, Montes S, et al. Infección por adenovirus en hospital de niños con enfermedades respiratorias crónicas. Rev Chil Pediatr 2013; 84 (5): 522-6. https://scielo.conicyt.cl/pdf/rcp/v84n5/art06.pdf [ Links ]
21.- Henrickson KJ. Cost-effective use of rapid diagnostic techniques in the treatment and prevention of viral respiratory infections. Pediatr Ann 2005; 34 (1): 24-31. PMID: 15693213. [ Links ]
22.- Gerber S I, Erdman D D, Pur S L, Díaz P S, Segreti J, Kajon A E, et al. Outbreak of adenovirus genome type 7d2 infection in a pediatric chronic-care facility and tertiary-care hospital. Clin Infect Dis 2001; 32 (5): 694-700. DOI: 10.1086/319210. [ Links ]
23.- Larrañaga C, Vicente M, Wu E, Carrasco L, Pena A, Onate C, et al. Adenovirus in children with acute lower respiratory tract infections. Rev Chil Pediatr 1988; 59 (5): 312-7. PMID: 2856008. [ Links ]
24.- Levy Y, Nitzan M, Beharab A, Zeharia A, Schoenfeld T, Nutman J, et al. Adenovirus type 3 infection with systemic manifestation in apparently normal children. Isr J Med Sci 1986; 22 (11): 774-8. PMID: 3025130. [ Links ]
25.- Martin ET, Kuypers J, McRoberts J P, Englund J A, Zerr D M. Human bocavirus 1 primary infection and shedding in infants. J Infect Dis 2015; 212 (4): 516-24. DOI: 10.1093/infdis/jiv044. [ Links ]
26.- Broccolo F, Falcone V, Esposito S, Toniolo A. Human bocaviruses: Possible etiologic role in respiratory infection. J Clin Virol 2015; 72: 7581. DOI: 10.1016/j.jcv.2015.09.008. [ Links ]
27.- Harada Y, Kinoshita F, Yoshida L M, Minh le N, Suzuki M, Morimoto K, et al. Does respiratory virus coinfection increases the clinical severity of acute respiratory infection among children infected with respiratory syncytial virus? Pediatr Infect Dis J 2013; 32 (5): 441-5. DOI: 10.1097/INF.0b013e31828ba08c. [ Links ]
28.- Jiang W, Yin F, Zhou W, Yan Y, Ji W. Clinical significance of different virus load of human bocavirus in patients with lower respiratory tract infection. Sci Rep 2016; 6: 20246. DOI: 10.1038/srep20246. [ Links ]
29.- Moyer K, Wang H, Salamon D, Leber A, Mejias A. Enterovirus D68 in hospitalized children: sequence variation, viral loads and clinical outcomes. PLoS One 2016; 11 (11): e0167111. https://doi.org/10.1371/journal.pone.0167111. [ Links ]
Received: July 09, 2018; Accepted: December 19, 2018
Correspondencia a: Loreto Fuenzalida Inostroza lfuenzalidai@uautonoma.cl
No existe conflicto de interés en los autores.
This is an Open Access article distributed under the terms of the Creative Commons Attribution Non-Commercial License, which permits unrestricted non-commercial use, distribution, and reproduction in any medium, provided the original work is properly cited.






