HLA DRB1, DQB1, DPA1* y DPB1* y su asociación con la patogénesis de las leucemias en población venezolana
Sergio E. Rivera-Pirela,(1) Miriam Echeverría,(1) Pedro Salcedo,(2) Georgina Márquez,(2) Zuhey Carrillo,(1) Yennis Parra,(1) Ana María Cipriani,(1) José R. Núñez,(1) Melchor Álvarez de Mon (3)
(1) Universidad del Zulia, Faculta de Medicina, División de Investigación. Maracaibo, Zulia, Venezuela • (2) Instituto Hematológico de Occidente y
Banco de Sangre del Estado Zulia, Laboratorio HLA e Inmunología. Maracaibo, Zulia, Venezuela • (3) Universidad de Alcalá de Henares, Departamento de Medicina. Madrid, España
Correspondencia: Sergio E. Rivera-Pirela. sergio.rivera54@hotmail.es
How to cite this article: Rivera-Pirela SE, Echeverría M, Salcedo P, Márquez G, Carrillo Z, Parra Y, Cipriani AM, Núñez JR, Álvarez de Mon M. [HLA DRB1*, DQB1*, DPA1*, and DPB1* and their association with the pathogenesis of leukemia in the population of Venezuela]. Rev Alerg Mex. 2016 Jul-Sep;63(3):237-251.
Background: The HLA complex is involved in the pathogenesis of leukemia.
Objectives: The presence of class II HLA alleles DRB1 *, DQB1 *, DPA1*, and DPB1 * was evaluated in 47 patients with acute lymphoblastic leukemia (ALL) and 48 with chronic myeloid leukemia (CML) for comparison with 48 healthy volunteers in Zulia, Venezuela, and to evaluate potential associations of HLA with leukemia.
Methods: Low- and high-resolution PCR-SSP was used for class II HLA regions DRB1 *, DQB1 *, DPA1 *, and DPB1 * following the instructions of KIT Olerup SSP Genovision.
Results: Alleles HLA-DRB1*14, especially DRB1*14:21, -DPA1*1:06, -DPA1*01:03,-DPA1*02:01, and the haplotypes HLA-DPA1*01:03- DPB1*04:01, DPA1*01:03-DPB1*02:01, DPA1*01:03-DPB1*99:01, -DRB1*14-DPA1*01:03, -DRB1*15-DPA1*01:03 were associated with CML (RR > 3); alleles HLA-DRB1*13, -DQB1*02, -DPA1*01:05, -DPA1*01:09 and the haplotypes HLA-DPA1*01:09-DPB1*02:01, DPA1*01:09- DPB1*04:01 were protective (RR < 1). Alleles HLA-DQB1*04, -DQB1*05, -DPA1*1:06, -DPA1*01:07, -DPA1*1:08 had a positive association with ALL. Alleles HLA-DPA1*01:09, -DPA1*02:01, -DPB1*02:01, -DPB1*03:01 and the haplotypes HLA-DPA1*01:03-DPB1*04:02, -DPA1*01:09- DPB1*02:01, -DPA1*01:09-DPB1*04:01, -DPA1*02:01-DPB1*04:02 were negatively associated.
Conclusions: The absence of associations with HLA-DRB1 * region in ALL and other association patterns identified suggest marked differences in the pathogenesis of leukemia, which suggests possible deficiencies in antigen presentation for ALL or potential effects of molecular mimicry in CML.
Keywords: Human leukocyte antigens; Acute lymphocytic leukemia; Chronic myelogenous Leukemia; Venozolan mestizo population.
Este artículo debe citarse como: Rivera-Pirela SE, Echeverría M, Salcedo P, Márquez G, Carrillo Z, Parra Y, Cipriani AM, Núñez JR, Álvarez de Mon M. HLA DRB1*, DQB1*, DPA1* y DPB1* y su asociación con la patogénesis de las leucemias en población venezolana. Rev Alerg Mex. 2016;63(3):237-251.
Resumen
Antecedentes: El complejo de histocompatibilidad es un factor que influye en la patogénesis de las leucemias.
Objetivos: Se evaluó la presencia de alelos HLA clase II DRB1*, DQB1*, DPA1* y DPB1* en 47 pacientes con leucemia linfoide aguda (LLA) y 48 con leucemia mieloide crónica (LMC), para compararlos con 48 voluntarios sanos de Zulia, Venezuela, y determinar las posibles asociaciones de HLA con las leucemias.
Métodos: Se utilizó la técnica de PCR-SSP de baja y alta resolución para las regiones HLA clase II DRB1*, DQB1*, DPA1* y DPB1* conforme las instrucciones del KIT Olerup SSP Genovision.
Resultados: Los alelos HLA-DRB1*14, especialmente DRB1*14:21, -DPA1*1:06, -DPA1*01:03,-DPA1*02:01, y los haplotipos HLA-DPA1*01:03- DPB1*04:01, DPA1*01:03-DPB1*02:01,DPA1*01:03-DPB1*99:01, -DRB1*14-DPA1*01:03, -DRB1*15-DPA1*01:03 tuvieron asociación con LMC (RR > 3); los alelos HLADRB1* 13, -DQB1*02, -DPA1*01:05, -DPA1*01:09 y los haplotipos HLA-DPA1*01:09-DPB1*02:01, DPA1*01:09- DPB1*04:01 resultaron protectores (RR < 1). Los alelos HLA-DQB1*04, -DQB1*05, -DPA1*1:06, -DPA1*01:07, -DPA1*1:08 tuvieron asociación positiva con LLA. Los alelos HLA-DPA1*01:09, -DPA1*02:01, -DPB1*02:01, -DPB1*03:01 y los haplotipos HLA-DPA1*01:03- DPB1*04:02, -DPA1*01:09-DPB1*02:01, -DPA1*01:09- DPB1*04:01, -DPA1*02:01-DPB1*04:02 resultaron asociados negativamente.
Conclusiones: La ausencia de asociaciones con la región HLA-DRB1* en LLA y los otros patrones de asociación identificados sugieren marcadas diferencias en las patogénesis de las leucemias, lo que orienta hacia posibles deficiencias en la presentación antigénica para LLA o posibles efectos de mimetismo molecular en LMC.
Palabras clave: Antígenos leucocitarios humanos; Leucemia linfoide aguda; Leucemia mieloide crónica; Población mestiza venezolana.
Abreviaturas y siglas
DNA, deoxyribonucleic acid
FONACIT, Fondo Nacional para la Ciencia y la Tecnología
HLA, antígenos leucocitarios humanos
LLA, leucemia linfoide aguda
LLC, leucemia linfoide crónica
LMC, leucemia mieloide crónica
mRNA, messenger RNA
pH, potencial de hidrogeniones
RR, riesgo relativo
SSP-PCR, single specific primer-polymerase chain reaction
Introducción
Las enfermedades hemato-oncológicas tienen gran relevancia debido a la elevada incidencia de mortalidad en la población infantil y adulta joven.1 Tal es el caso de las leucemias, de las cuales se conoce que para el año 2000 unas 256 000 personas en 14 regiones diversas del mundo desarrollaron algún tipo de leucemia mostrando un 82% de mortalidad.2 Las leucemias son más frecuentes en hombres (56%) que en mujeres y la incidencia varía de acuerdo con la edad y tipo de leucemia, aun cuando ocupan el décimo lugar de morbilidad.
En Norteamérica representan el 25% de todos los cánceres en niños menores de 20 años en el período 1997-2002, y la leucemia linfoide aguda (LLA) es la más frecuente en este grupo de edad. En México, en el año 2002, ocupó el segundo lugar en mortalidad por neoplasias malignas en niños de 5 a 14 años de edad, con una tasa de 2.71 por 100 000 habitantes.1 Las LMC representan entre el 15 y el 20% del total de leucemias y su incidencia en los países occidentales se estima en 1.6 casos por 100 000 habitantes/año.
La edad mediana de su aparición es de 53 años, la incidencia máxima se encuentra entre los 40 y los 60 años. Predomina ligeramente en varones, con una relación de 1.3:1. Alrededor de la mitad de los pacientes son asintomáticos al diagnóstico y la mediana de supervivencia global es de 4 a 6 años.3
En el caso de Venezuela para el año 2006 las leucemias ocupaban el quinto lugar entre las diez principales localizaciones de cáncer en hombres y mujeres. En niños y adolescentes menores de 15 años representa, junto con las neoplasias malignas del sistema nervioso central y los linfomas, más del 40% de la patología neoplásica infantil, donde solo el 30 % es susceptible de trasplante.4
Se ha señalado, que la genética poblacional tiene un gran impacto en la epidemiología de las poblaciones, y eso puede ser una extraordinaria herramienta para el entendimiento del estado de salud, la demografía, la historia y la antropología si se considera que, étnicamente, las poblaciones mezcladas pueden ser analizadas por regiones dentro de su propio país.5
Las cifras epidemiológicas presentadas anteriormente para Venezuela motivan a dirigir la atención en la investigación de leucemia en esta población debido, principalmente, a que en estudios previos sobre antígenos de histocompatibilidad realizados por Villalobos y colaboradores, 6 en una población mestiza venezolana, se observó un incremento significativo en la frecuencia de los alelos HLA-B*39 y HLA-C*03 en pacientes con leucemia mieloide crónica (LMC). Varios haplotipos HLA clase I de dos loci, mostraron diferencias significativas en estos pacientes LMC con respecto a los controles. El haplotipo de tres loci HLA-A*02, B*40, C*03 se asoció positivamente a la LMC. En este estudio no se encontró ningún tipo de asociación con leucemia linfoide aguda.
Se realizó la tipificación de los alelos de HLA DRB*1, DQB1*, DPA1* y DPB1* en pacientes con leucemia y controles sanos para evaluar el grado de asociación HLA en estos pacientes venezolanos, como un aporte en la compresión del origen y patogénesis de esta mortal enfermedad.
Métodos
La población de estudio estuvo conformada por 95 pacientes leucémicos y 48 individuos mestizos venezolanos de cualquier sexo, con al menos tres generaciones nacidas en el país, aparentemente sanos. Esta se clasificó de la siguiente manera: 47 pacientes con LLA en edades comprendidas entre 7 meses a 65 años; 48 pacientes con LMC en edades entre 18 a 70 años y 48 individuos sanos sin evidencia clínica de enfermedad autoinmune o tumoral en edades comprendidas entre 8 a 78 años.
Los pacientes fueron seleccionados previo diagnóstico de LLA y LMC, el cual se realizó a través de exámenes de citogenética, citometría de flujo y revisión de historias clínicas, entre quienes asistieron de manera voluntaria a las consultas de Hematología de los Hospitales de Especialidades Pediátricas del Instituto Venezolano de los Seguros Sociales Dr. Adolfo Pons y Central Dr. Urquinaona, ubicados en el municipio Maracaibo, estado Zulia, Venezuela.
Tanto los pacientes como los individuos sanos fueron sometidos a un cuestionario-encuesta que sirvió para evaluar el estado de salud general y los antecedentes familiares sobre enfermedades. Todos los individuos en el estudio fueron debidamente notificados acerca del objetivo e implicaciones de participar en este y se les pidió firmar de manera voluntaria un consentimiento informado.
Todos los procedimientos realizados se rigieron por lo establecido en la Declaración de Helsinki7 y en el Código de Bioética del Fondo Nacional para la Ciencia y la Tecnología de Venezuela (FONACIT)8 para procedimientos experimentales en humanos. La tipificación de los alelos HLA, que involucró la extracción y amplificación del DNA se realizó en el Laboratorio de HLA e Inmunología del Instituto Hematológico de Occidente (Banco de Sangre) de la ciudad de Maracaibo-estado Zulia.
La concentración de los DNA se midió en el Laboratorio de Neurociencias del Instituto de Investigaciones Biológicas de la Facultad de Medicina de la Universidad del Zulia. Para el estudio se excluyeron aquellos pacientes con LLA y LMC que habían recibido transfusiones de sangre recientes, al igual que los individuos aparentemente sanos con diagnóstico de alguna patología degenerativa, neoplásica, procesos infecciosos y trastornos hematológicos.
Luego de ayuno nocturno (mínimo de 8 horas) y bajo condiciones de asepsia, a todos los sujetos se les extrajo DNA genómico a partir de sangre total mediante la técnica de precipitación de sales.9 La amplificación genética se realizó con la técnica de PCR-SSP (reacción en cadena de la polimerasa) y se usaron cebadores con secuencias específicos de la región amplificar.
Los alelos HLA de clase II (DRB1*, DQB1*, DPA1* y DPB1*) se analizaron con la técnica de PCR-SSP, Olerup SSP, (Genovision) DRB1*-DQB1*, DPA1* y DPB1* de los respectivos lotes (Lot No x 80.73G, 47G), lo cual permitió evaluar 277 alelos del locus HLA DRB1*, 3*,4*, 34 alelos del locus DQB1*, 28 alelos del locus DPA1*, 123 alelos DPB1* y 64 subtipos del alelo DRB1*14. Para este último, se incluyeron solamente ocho pacientes del total de 21 con LMC y cuatro controles que resultaron positivos al alelo DRB1*14 asociados positivamente con la LMC. Estos subtipos se determinaron utilizando el kit de PCR-SSP Genovision Nº de lote 01.117-12/03-withhout Taq –polimerasa de alta resolución.
Las frecuencias alélicas y genotípicas se determinaron empleando el paquete de análisis genético Genepop.10 El equilibrio de Hardy Weinberg para cada locus se probó con el paquete Arlequín, versión 3.0, el cual emplea una prueba exacta, análoga a la prueba Fisher, que toma en cuenta modificaciones al algoritmo de la cadena de Markov, descrita por Guo y Thompson en 1992.11 Las frecuencias haplotípicas fueron estimadas igualmente en Arlequín versión 3.0. Las pruebas de asociación se realizaron utilizando el programa SAS con el procedimiento Prim Comp. Se utilizó un intervalo de confianza del 95% y el valor de alfa fue < 0.05. La asociación HLA LLA o LMC se consideró positiva si el riesgo relativo (RR) era > 3 y negativa si RR era < 1. Los datos obtenidos de la determinación de alelos se registraron utilizando el paquete estadístico SPSS versión 17.0 en una base de datos computarizada (Sandiego, CA, USA) y el programa Excel para Windows 2007 para cálculo y presentación de datos en los cuadros y gráficos.
Resultados
Al comparar las frecuencias de los alelos HLA-DRB1* en pacientes con leucemia linfoide aguda e individuos sanos, no se observaron asociaciones positivas o negativas que pudieran determinar la presencia de alelos de protección ni de susceptibilidad asociados a la LLA.
Los estudios estadísticos demostraron que el alelo DQB1*04 (RR= 5.1; IC95% < 0.05) resultó asociado positivamente con la LLA y el DQB1*05 (RR= 0.36; IC95% <0.05) negativamente (Cuadro 1).
Cuadro 1. Asociación entre HLA DQB1* y LLA.

Los alelos DPA1*01:07 (RR= 24.1; IC95% <0.0001), DPA1*01:08 (RR=22; IC95% <0.0001), DPA1* 01:06 (RR 14.87: IC95% <0.01) resultaron asociados positivamente con la LLA. El ale o DPA1*01:09 presentó un RR de 0.04 con una IC95% <0.0001 y el RR para el alelo DPA1*02:01 fue de 0.22, IC95% <0.0001, y resultaron asociados negativamente a la LLA (Cuadro 2).
Cuadro 2. Asociación entre HLA DPA1* y LLA

Respecto al locus DPB1* se encontraron solo asociaciones negativas con los alelos DPB1* 02:01 (RR= 0.2; IC95% <0.01) y DPB1*03:01 (RR=0.15. IC95% < 0.0025) y las LLA (Cuadro 3).
Cuadro 3. Asociación entre HLA DPB1* y LLA
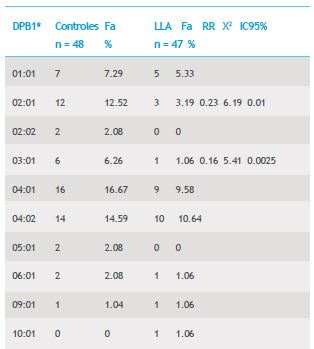


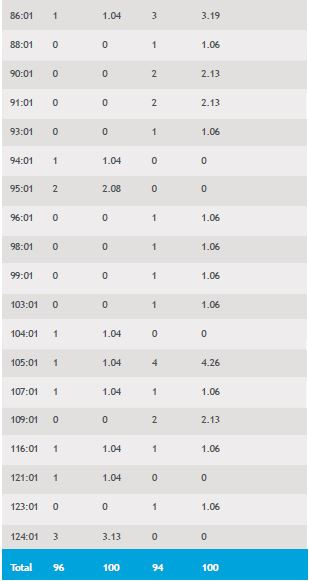
En la combinación de haplotipos HLA-DRB1*/DPA1* solo se evidenció una asociación negativa con el haplotipo DRB1*04/DPA1*02:01 (RR=0.09 y IC95% < 0.005) la cual estuvo presente en nueve controles y en un solo paciente con leucemia linfoide aguda. Los haplotipos HLA-DPA1*/DPB1*01:03-04:02 y 02:01-04:02 resultaron asociados negativamente con las LLA, no observándose ninguna asociación positiva con la enfermedad.
El alelo DRB1*14 resultó el de mayor frecuencia para la región DRB1* con un RR=13.8; IC95% <0.0001, asociado positivamente con las LMC, constituyéndose en un alelo de riesgo para esta patología. El alelo DRB1*13, mostró una asociación negativa, con RR=0.35, IC95% <0.01 identificándose como un alelo de protección para la LMC (Cuadro 4).
Cuadro 4. Asociación entre HLA DRB1* y LMC
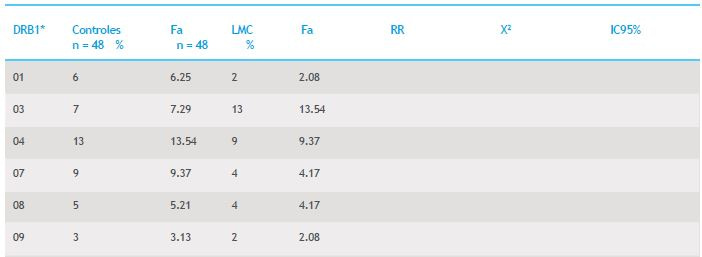

Los resultados de la evaluación de los subtipos HLA-DRB1*14 en ocho pacientes con LMC y cuatro controles sanos, demostraron que el 59.09% de los pacientes con LMC presentan el subtipo HLADRB1* 14:21, mientras que en los controles mestizos venezolanos los subtipos más frecuentes fueron DRB1*14:22, DRB1*14:34 y DRB1*14:52 con 25% cada uno (Figura 1).
Figura 1. Frecuencia de subtipos del HLA-DRB1*14 determinados por PCR-SSP de alta resolución Genovision en controles sanos y pacientes
con LMC.

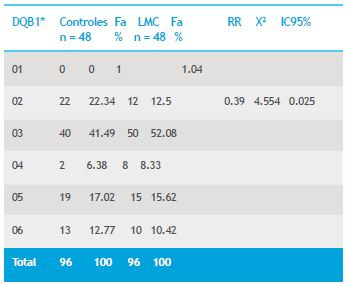
Los controles HLA-DRB4* mostraron un porcentaje de homocigocis del 54.54%, seguido de los pacientes LLA con 52.38% y los LMC con 42.85%. Se pudo observar una asociación negativa del alelo DQB1*02 y la LMC (Cuadro 5).
Cuadro 5. Asociación entre HLA DQB1* y LMC
En los pacientes LMC mestizos venezolanos se observaron 6 pacientes Ph+ y 7 Ph-. En la serie evaluada, 3/6 pacientes LMC Ph+ resultaron DRB1*14 positivos y 3/6 Ph + no mostraron este alelo. Sin embargo 6/7 Ph- fueron igualmente DRB1*14 negativos y mostraron un ligero paralelismo entre la presencia del cromosoma Ph y el alelo LMC asociado positivo DRB1*14. Se hace necesario aumentar la serie de pacientes para poder corroborar esta observación.
Al comparar la frecuencia de alelos entre los controles con los pacientes LMC se encontró una asociación positiva del HLA-DPA1*01:03 con (RR=8.6, IC95% < 0.0001) y el HLA-DPA1* 02:01 (RR=3.66, IC95% < 0.001). Los alelos DPA1*01:05 con un (RR= 0.03, IC95% <0.0001) y el DPA1*01:09 con un (RR=0.01, IC95% < 0.0001), mostraron una asociación negativa (Cuadro 6). En la región DPB1*- no se observó asociación positiva ni negativa con la LMC.
Cuadro 6. Asociación entre HLA DPA1* y LMC

Los resultados de la comparación de los haplotipos DRB1*/DPA1* demuestran que existen por lo menos dos asociaciones positivas de estos haplotipos con las LMC, DRB1*/DPA1*14/01:03 (RR= 12.6, IC95% < 0.0005) y 15/01:03 (RR= 13, IC95% < 0.025). El haplotipo DPA1*01:03/DPB1*04:01(RR=3.26, IC95% < 0.05) resultó de riesgo, entretanto, los haplotipos DPA1*01:09-DPB1* 02:01(RR=0.07, IC95% < 0.01) y DPA1*01:09- DPB1* 04:01(RR= 0.07, IC95% < 0.01) resultaron asociados negativamente a las LMC.
Discusión
Al aumentar la serie de pacientes y controles de las LLA y LMC en venezolanos, se pudieron confirmar las asociaciones positiva de LMC con el HLA-DRB1*14 y negativa con el -DRB1*13. El subtipo predominante fue el alelo DRB1*14:21 con un 59.09% en pacientes LMC, totalmente ausente en los controles por lo que podemos considerarlo como una marcador para LMC en esta población. Llama la atención que en esta serie no se observó ninguna asociación positiva o negativa del HLA-DRB1* con las LLA en venezolanos. Esto marca una gran diferencia entre ambas patologías.
Casi todos los pacientes LMC Ph+ positivos expresan transcripciones de fusión b3a2 o b2a2, dependiendo de si está incluido el exón b3. Cuando traduce, b2a2 y b3a2 cada RNAm genera una proteína BCR-ABL de 210 kDa (p210), que es necesaria y suficiente para la transformación leucogénica.12 En los pacientes LMC venezolanos se observaron 6 Ph+ y 7 Ph-. En la serie evaluada, 3/6 pacientes LMC Ph+ resultaron DRB1*14 positivos y 3/6 Ph+ no mostraron este alelo. Sin embargo 6/7 Ph- fueron igualmente DRB1*14 negativos y mostraron un ligero paralelismo entre la presencia del cromosoma Ph y el alelo DRB1*14 asociado positivamente la LMC.
La asociación positiva de los alelos HLA con los transcriptos de fusión b3a2 o b2a2 sugiere que ese alelo en particular no es eficiente en el reconocimiento y la presentación del péptido derivado de un transcripto portador de la p120 bcr-abl, por lo tanto serán deficientes en la activación de células T citotóxicas capaces de eliminar las células leucémicas defectuosas. Yasukawa et al.13 reportan en pacientes japoneses con LMC una asociación positiva HLA-DRB1*12:01 con b2a2 y -DRB1*04:03, -DRB1*08:02,-DRB1*14:03, -DRB1*14:05 con b3a2; igualmente, una asociación negativa -DRB1*04:05, -DRB1*08:03:02, -DRB1*15:02, con b2a2 and -DRB1*08:03:02, -DRB1* 15:01 con b3a2.14
Los controles HLA-DRB4* mostraron un porcentaje de homocigocis del 54.54%, seguido de los pacientes LLA con 52.38% y los LMC con 42.85% en pacientes venezolanos. La asociación predisponente del DR53 (DRB4*) y la LLC publicada por orak et al.15-19 fue replicada en los pacientes LLC en US con el DRB4*01:01 mientras que los pertenecientes al protector DR52 (DRB3) asociado a LLC, correspondieron a los haplotipos HLA clase II DRB3*03:01; DRB1*13:02; DQB1*06:04, DRB3*02:02; DRB1*13:01; DQB1*06:03 y DRB3*02; DRB1*11; DQB1*03. Se observó un haplotipo DRB3* predisponente, el DRB3*02; DRB1*11; DQB1*06. El haplotipo DRB4* muestra varios alelos predisponentes en esta población tales como los alelos observados en familias DRB1*04, DRB1*07 y DRB1*09.20 Solamente el DRB1*13 coincide como protector asociado negativamente a la LMC en pacientes venezolanos concordando con lo observado en pacientes LLC de Estados Unidos.20
Una fuerte asociación HLA-DQB1*0201, -DQB1*0302, -DQB1*0303 y -DRB1*1302 fue reportada en 824 casos de pacientes LLA-BCP confrontados con 4.737 controles en la población del Reino Unido.21,22 En pacientes mexicanos los alelos HLA DRB1*01:02, DQB1*02:01/02 y DPB1*04:01 resultaron protectores para la LLA, el alelo DQB1*02 resultó protector también para las LMC en venezolanos. DQB1*03:01 y DPB1*04:02 se reportaron como de riesgo para las LLA en dicha población mexicana. Indistintamente, estos resultados difieren de los obtenidos en nuestra serie de pacientes LLA.23
En pacientes caucásicos de Estados Unidos con leucemia linfoide crónica (LLC) se describen los alelos HLADQB1* 05:04 asociados positivamente mientras que el alelo DRB1*04:03 se encuentra asociado negativamente y actúa como protector en las LLC en dicha población; el alelo DRB1*09:01 se encuentra también asociado a las LLC en pacientes afroamericanos.20 En correspondencia con lo anterior, el haplotipo DRB1*04/DPA1*02:01 se asoció negativamente a las LLA en venezolanos.
El haplotipo DRB4*01:01/DRB1*07:01/DQB1*03:03 se encuentra universalmente asociado positivamente a LLC en pacientes blancos, afroamericanos e hispanos. 20 Los pacientes LLC judíos ashkenazi, muestran asociación positiva con los alelos DRB4*01:01, DRB1*04:02 y DQB1*03:02.20 Por el contrario, el alelo DQB1*04 resultó asociado positivamente con la LLA y el DQB1*05 negativamente en pacientes LLA venezolanos, mientras que el DQB1*02 aparece asociado negativamente a la LMC.
El alelo HLA-DPA1*01:06 muestra una asociación positiva con LLA y LMC en venezolanos por lo que pudiera considerarse como un marcador de leucemias en esta población. Los alelos DPA1* asociados a LLA, entre los ya publicados24 y los nuevos, posterior al incremento de la serie de pacientes son DPA1*01:03,02:01(nuevos), -DPA1*01:07 y -DPA1*01:08 LLA (confirmados).24 Los haplotipos HLA-DRB1*14-DPA1*01:03 y HLA-DRB1*15- DPA1*01:03 resultaron incrementados significativamente en pacientes LMC venezolanos. Poca o ninguna información se logró recopilar sobre asociación de las leucemias y la región HLA-DPA1*. Previamente habíamos publicado los diferentes patrones de distribución de los genotipos PA1* en las LLA y LMC.
El genotipo DPA1*01:03:01- 01:03:02/02:01:01-02:02:06 muestra un 40.4% en controles, 8.5% en LLA y 64.6% en LMC, aunado a que en las LMC se observan muy pocos alelos DPA1*.24 Más adelante ahondaremos en las posibles implicaciones de estos resultados en la patogénesis de las leucemias.
El alelo HLA-DPB1*99:01 resultó asociado positivamente, sin confirmación de Yate, a las LMC. Ninguna asociación negativa se pudo observar entre alelos de la región DPB1* y las LMC. Sin embargo, el haplotipo HLA-DPA1*01:03-DPB1-*99:01, conjuntamente con el -DPA1*01:03-DPB1*02.01 y el -DPA1*01:03-DPB1*04:0, resultaron asociados positivamente; mientras que los haplotipos HLA-DPA1*01:09-DPB1*02:01 y -DPA1*01:09- DPB1*04:01 se mostraron asociados negativamente a las LMC.
Solo se obtuvieron asociaciones negativas entre alelos DPB1* y la LLA, especialmente con -DPB1*02:01 y -DPB1*03:01 así como con los haplotipos DPA1*01:03- DPB1*04:02, DPA1*01:09-DPB1*02:01, DPA1*01:09-DPB1* 04:01 y DPA1*02:01-DPB1*04:02, también asociados negativamente.
Estos resultados confirman todas las asociaciones DRB1*,DQB1* y DPA1* previamente publicadas. Nuevos alelos aparecieron asociados al aumentar la serie de pacientes sin observarse resultados que pudiesen cambiar las conclusiones del estudio preliminar, por el contrario fianzamos las mismas y aportamos nuevas perspectivas en referencia a la posible participación de los alelos HLA en la patogénesis de la enfermedad.25
Las asociaciones positivas o negativas de alelos del sistema HLA con el desarrollo de enfermedades es un hecho patogénico establecido. Esta asociación se ha demostrado en entidades nosológicas de muy diferente naturaleza incluyendo procesos autoinmunes/inflamatorios, infecciosos y neoplásicos. La consistencia de estas asociaciones es diferente entre distintas razas y etnias.
En la serie de pacientes con LLA no se observaron asociaciones positivas ni negativas con la región HLA-DRB1*, lo cual marcó una importante diferencia con las LMC que se suma a los diferentes patrones de distribución observados para ambas leucemias de la región DP conformada por los alelos DPA1* y DPB1*. Las frecuencias alélicas de región DR en los pacientes con LLA no muestran diferencias con las mostradas por los controles. Por el contrario, la región DPA muestra una fuerte diferencia con la distribución alélica de los controles, la cual es menos marcada en pacientes LMC. En estos últimos, tampoco se observan diferencias marcadas con los controles en referencia a las frecuencias DPB. Estos resultados pudieran estar indicando que los pacientes con LLA adolecen de una cantidad de alelos protectores que pudieran estar implicados en una conveniente presentación antigénica a las células inmunocompetentes.
Refiere la literatura que las variaciones observadas en la asociación de la región DPB1* con las leucemias se debe a diferentes presiones de selección ejercidas por diversos patógenos específicos de cada región. La molécula HLA-DPB1*06:01 pudiera estar funcionalmente implicada en la leucemia infantil.26 De ahí el interés en esta región estrechamente relacionada con la patogénesis de las leucemias.27 La exposición a infecciones comunes y el papel de procesos inmunes relacionados la postulan como un fuerte factor de riesgo de las LLA en niños. El reconocimiento por parte de las células T de complejos DP1-péptido en el contexto de una respuesta inmunitaria insuficientemente modulada, pudiera generar una respuesta desfavorable capaz de contribuir con la leuquemogénesis.28 La patogénesis de la LMC, por el contrario, pudiera estar relacionada preferiblemente con mimetismo molecular de las secuencias peptídicas de los antígenos HLA fuertemente asociados a esta enfermedad como blanco de ataque sistemático de ciertos patógenos o deficiencias en la regulación de las poblaciones linfocitarias encargadas de eliminar las células cancerígenas. En leucemia mieloide crónica, las asociaciones positivas con el DRB1*14 y su implicación en el mimetismo molecular con ciertos adenovirus sugerida por Dorak et al. refuerza la hipótesis de que estos pacientes pudieran estar desarrollando respuestas autorreactivas contra dichas moléculas inducen la leuquemogénesis. Por tal motivo se propone continuar investigando la posible presencia de células T autorreactivas en pacientes con LMC capaces de reconocer además secuencias peptídicas del adenovirus posiblemente implicado en la patogénesis de este cuadro leucémico.
Agradecimientos
La investigación que dio origen a este artículo fue subvencionada por el Proyecto CONDES No. CC-0187-11 y el Instituto Hematológico de Occidente, Maracaibo, Estado Zulia, Venezuela.
Referencias Bibliográficas
1. Tirado-Gomez LL, Mohar Betancourt A. Epidemiología de las neoplasias hemato-oncológicas. Cancerologia. 2007;2:109-20. [Citado 2015 May 03]. Disponible en: http://www.incan.org.mx/revistaincan/elementos/ documentosPortada/1193426695.pdf
2. World Health Organization [Internet]. Colin DM, Boschi-Pinto C, Lopez AD, Murray CJL. Cancer incidence, mortality and survival by site for 14 regions of the world. Global Programme on Evidence for Health Policy Discussion Paper No. 13. 2001. [Citado 2014 Oct 10] Disponible en: http://www.who.int/entity/healthinfo/paper13.pdf
3. Rodríguez M, Cardona AF, Grajales MA, Enciso L, Ruiz G, Yepes A, et al. Leucemia mieloide crónica en crisis blástica bases moleculares y diagnóstico. Rev Venez Oncol. [Internet]. 2007; 19(4):287-296 [Citado 2016 Jul 02]. Disponible en: http://www.scielo.org.ve/scielo.php?script= sci_arttext&pid=S0798-05822007000400003&lng=es.
4. Capote Negrin LG. Aspectos epidemiológicos del cáncer en Venezuela. Rev. venez. oncol. [Internet]. 2006;18(4):269-281 [Citado 2016 Jul 02]. Disponible en: http://www.scielo.org.ve/scielo.php?script=sci_arttext&pid=S0798-05822006000400011&lng=e
5. Gorodezky C. Genetic difference between Europeans and Indians. Allergy Proc. 1992;13(5):243-250.
6. Villalobos C, Rivera S, Weir-Medina J, Hassanhi M, Montiel M, Gonzales R. Asociación HLA clase I y leucemia en pacientes mestizos del estado Zulia, Venezuela.. Invest Clin. 2003;44(4):283-289.
7. Ramiro GFJ. Declaración de Helsinki de la Asociación Médica Mundial. Principios éticos para las investigaciones médicas en seres humanos. 2005. Disponible en: http://www.bioeticaweb.com/index2.php?option= com_content&do_pdf=1&id=896 (citado en abril del 2011).
8. Briceño E, Suárez E, Michelangeli C, Feliciangeli D, Otaiza E, Mendible JC, et al. Código de biotética y bioseguridad. 2ª. edición. Venezuela: Ministerio de Ciencia y Tecnología (FONACIT); 2002. (Citado 2011 Feb 20). Disponible en: http://www.miproyecto.gov.ve/anexos/bioetica.pdf
9. Miller SA, Dkyes DD, Polesky HF. A simple salting out procedure for extracting DNA from human cell. Nucleic Acids Res. 1988;16(3):1215.
10. Raymond M, Rousset F, GENEPOP (version 1.2) population genetics software for exact test and ecumerism. J Herad. 1995;86:248-249.
11. Excoffier L, Laval G, Schneider S. Arlequin ver 3.0: An integrated software package for population genetics data analysis. Evol Bioinform Online 2005;1:47-50.
12. McLaughlin J, Chianese E, Wittte ON. In vitro transformation of immature hematopoietic cells by the p210 bcr/abl oncogen product of the Philadelphia chromosome. Proc NatlAcad Sci USA. 1987;84:6558-6562.
13. Yasukawa M, Ohminami H, Kojima K, Inokuchi K, Nishimura Y, Fujita S. Analysis of HLA-DRB1 alleles in Japanese patients with chronic myelogenous leukemia. Am J Hematol 2000;63(2):99-101.
14. Yasukawa M, Ohminami H, Kojima K, Hato T, Hasegawa A, Takahashi T, et al. HLA class II-restred antigen presentation of endogenus bcr-abl fusion protein by chronic myelogenus leukemia-derived dendritic cells to CD4+ T lymphocytes. Blood. 2001;98(5):1498-1505.
15. Dorak MT, Burnett AK. Molecular mimicry of an HLA-DR53 epitope by viruses [letter]. Immunol Today. 1994;15:138-139.
16. Dorak MT. The implications for childhood leukaemia of infection with adenovirus. Trends Microbiol. 1996;4:60-63.
17. Dorak MT, Lawson T, Machulla HKG, Darke C, Mills KI, Burnett AK. Unravelling an HLA-DR association in childhood acute lymphoblastic leukemia. Blood. 1999;94:694-700.
18. >Dorak MT, Oguz FS, Yalman N, Diler AS, Kalayoglu S, Anak S, Sargin D, Carin M. A male-specific increase in the HLA-DRB4(DR53) frequency in high-risk and relapsed childhood ALL. Leuk Res. 2002;26(7):651-656.
19. Dorak MT. MHC and leukemia. 2007. [Citado 2012 Ene 07]. Disponible en: http://www.dorak.info/mhc/mhcleuk.html
20. Gragert L, Fingerson S, Albrecht M, Maiers M, Kalaycio M, Hill BT. Fine-mapping of HLA associations with chronic lymphocytic leukemia in US populations Blood. 2014;124(17):2657-2665.
21. Hosking FJ, Leslie S, Dilthey A, Moutsianas L, Wang Y, Dobbins SE, et al. MHC variation and risk of childhood B-cell precursor acute lymphoblastic leukemia. Blood. 2011;117:1633-40. doi:10.1182/blood-2010-08-301598.
22. Kang H, Wilson CS, Harvey RC, Chen IM, Murphy MH, Atlas SR,, et al. Gene expression profiles predictive of outcome and age in infant acute lymphoblastic leukemia: a Children’s Oncology Group study. Blood. 2012;119(8):1872-81. doi: 10.1182/blood-2011-10-382861.
23. Godoresky C, Flores H, Alaez C, Sánchez D, Munguía A, Rodríguez A, et al. Spectrum of hla association with acute lymphoblastic leukemia in Mexican patients. Human Immunology. 2014;75 (Suppl):123.
24. Echeverría M, Rivera S, Márquez G, Carrillo, Z, Parra Y, Villalobos C. Patrones diferenciales de distribución de los alelos y genotipos HLADPA1* en asociación con leucemias linfoides agudas y leucemias mieloides crónica. Inmunologia. 2010;29(4):115-118.
25. Rivera PS, Echeverría M, Márquez G, Villalobos C, Pereira N, De Salvo L, et al. Asociación HLA-DR/DQ/DPA1 con leucemias linfoides agudas (LLA) y leucemias mieloides crónica (LMC) en la población mestiza del estado Zulia, Venezuela (estudio preliminar). Ciencia. 2007;15(4):365-372.
26. Wang XJ, Ai XF, Sun HY, Wang F, Xu SC, Wang ZQ. Relation of HLA-DRB1*15 with pathogenesis in 162 childhood cases of acute lymphoblastic leukemia. Zhongguo Shi Yan Xue Ye Xue Za Zhi. 2009;17:1507-1510.
27. Taylor GM, HussainA, Verhage V, et al. Strong association of the HLADP6 supertype with childhood leukaemia is due to a single allele, DPB1*0601. Leukemia. 2009;23:863-9. doi:10.1038/leu.2008.374.
28. Urayama K, Chokkalingam A, Metayer C, Ma X, Selvin S, Barcellos L, et al. HLA-DP genetic variation, proxies for early life immune modulation and Childhood acute lymphoblastic leukemia risk. Blood. 2012;120:3039-3047.