Evaluación de una técnica de PCR en tiempo real para determinar colonización por Streptococcus agalactiae en mujeres gestantes de Medellín que consultan en Dinámica IPS
Clara M. Duque1, Diana Marcela Sánchez1, Beatriz Gómez2, Jenny Andrea Carmona2, Damian Cifuentes3, Angela M. Gaviria1, Orville Hernández4
1 Grupo de investigación Biociencias IU Colegio Mayor de Antioquia Colombia
2 Dinamica IPS Colombia
3 Estudiante IU Colegio Mayor de Antioquia Colombia
4 Grupo de Investigación MICROBA. Escuela de Microbiología. Universidad de Antioquia Colombia
Calle 118 No. 15-24 Oficina 503, Bogotá, D. C., Colombia
Teléfono (57-1) 215 3714 y 215 3517
acin@etb.net.co
Resumen
Objetivo: Evaluar una técnica de PCR en tiempo real para determinar colonización por Streptococcus agalactiae en mujeres gestantes de Medellín.
Materiales y Métodos: Se realizó un estudio descriptivo prospectivo, en 150 mujeres gestantes, seleccionadas de forma aleatoria, en una IPS en el periodo comprendido entre Enero-Julio 2016. Criterio de inclusión: ser gestante entre la semana 35-37, declaración voluntaria de participación en el estudio y de exclusión el uso de antibióticos. A las pacientes, se les tomó muestra con hisopo del introito vaginal y de la región anal. Las muestras se procesaron para qPCR, cultivo en caldo selectivo con posterior siembra en agar sangre de carnero y medio cromogénico para S. agalactiae STRB (ChromIDTMStrepto, BioMérieuxSA.).
Resultados: La prevalencia de colonización por S. agalactiae en las gestantes fue de 20,9% y 22,3% en agar sangre y agar cromogénico STRB respectivamente, mientras que mediante PCR en tiempo real la prevalencia fue de 36%. Al comparar la qPCR con la prueba de oro se encontró: sensibilidad 79,31% (ICdel95%:0,61-0,90), especificidad 75,45% (IC del 95%: 0,66-0,82), valor predictivo positivo 46% (IC del 95%:0,32-0,59) y negativo 93,2% (IC del 95%: 0,86-0,96).
Discusión: El empleo de la qPCR permitió aumentar la sensibilidad y la oportunidad diagnostica (El tiempo requerido empleando el cultivo fue de 24-48 horas y por qPCR 6 horas), impactando la reducción de riesgos de transmisión neonatal de S. agalactiae, lo cual podría representar una disminución en días de estancia y costos hospitalarios por una infección prevenible.
Palabras clave: Streptococcus agalactiae, Cultivo, PCR en tiempo real, Colonización
Abstract
Objectives. To evaluate a real-time qPCR technique to determine colonization by Streptococcus agalactiae in pregnant women from Medellín.
Materials and Methods: A prospective and descriptive study was conducted in 150 pregnant women, randomly selected, at an IPS between January and July 2016. Inclusion criteria: gestation period week 35-37, voluntary declaration of participation in the study. Exclusion criteria: the use of antibiotics. Samples were taken from the vaginal introitus and the anal region using a hyssop and processed for qPCR as well as the gold standard test [selective broth culture with subsequent culture in blood agar and chromogenic medium for S. agalactiae STRB (ChromIDTMStrepto, BioMérieux SA)].
Results: The prevalence of colonization by S. agalactiae in pregnant women was 20.9% and 22.3% in blood agar and chromogenic agar STRB respectively, where as using qPCR the prevalence was 36.0%. The time required using the culture was 24-48h compared to 6h for qPCR. Our data comparing qPCR with the gold standard test showed: sensitivity 79.31% (95% CI: 0.61-0.90), specificity 75.45% (95% CI: 0.66-0.82), positive predictive value 46.0% (95% CI: 0.32-0.59) and negative 93.2%(95% CI: 0.86-0.96).
Discussion: The use of the qPCR increased the sensitivity and the diagnostic opportunity (4x to 8x faster using qPCR), which can lead to a decrease in the risk of neonatal transmission of S. agalactiae and result in a reduction in the length of hospital stay and costs induced by a preventable infection.
Key words: Streptococcus agalactiae, Real time PCR, Bacteriological Techniques
Introducción
Streptococcus agalactiae (S. agalactiae) es una causa importante de neumonía, sepsis y meningitis con alto índice de mortalidad en neonatos. La colonización se produce por la exposición del recién nacido al microorganismo durante el parto a partir del tracto genital materno colonizado, o en el útero por vía ascendente, siendo la tasa de transmisión vertical del 50%1-4. La importancia de realizar tamización de colonización de las gestantes antes del parto, radica en definir el empleo de antibióticos profilácticos para prevenir el desarrollo de la infección. El cultivo de muestras de región perianal e introito vaginal en caldo selectivo y posterior inoculación en Agar sangre es la prueba de oro para detectar colonización por S. agalactiae5. Sin embargo, los cultivos requieren varios días para obtener resultados y tomar las medidas profilácticas adecuadas.
Los ensayos moleculares basados en detección de ADN/ARN ofrecen herramientas prometedoras más sensibles, específicas y rápidas para identificación de S. agalactiae. Se han identificado en su genoma diversos blancos moleculares específicos, como el gen cfb, que codifica el factor CAMP6, el gen scpB la peptidasa C5a7 y el gen sip, una proteína inmunogénica de superficie 10,11.: Shabayek y col en 2010, emplearon PCR para la detección los genes cfb y scpB de Streptococcus grupo B con buenos resultados8. Rasoul-Yousefi-Mashouf y col 2014, emplearon la sub-unidad ribosomal 16S rRNA, de S. agalactiae para determinar la presencia de este microorganismo en muestras clínicas9. Mientras que Nabil Abdullah El Aila y col 2011 emplearon los genes sip y cfb en qPCR a partir de muestras clínicas10. Bergseng y col 2007 emplearon el gen sip11. Entre los estudios que emplean la qPCR se encuentran; el realizado en chicago en el cual la sensibilidad y especificidad fue del 95,8%, y del 64,5%17. Atkins y col en Washington reportaron una sensibilidad y especificidad del 86,8% y 95,2%14. Young y colaboradores en Boston una sensibilidad de 90,8%; y especificidad de 97,6%.18. Empleando PCR convencional, Fabien Rallu y col en Canadá, detectaron el gen scpB y cfb y reportaron una sensibilidad de 99,6%; y 75,3%; y especificidades de 100%, para ambos genes15.
Materiales y Métodos
Se realizó un estudio descriptivo prospectivo, se determinó una muestra a conveniencia de 150 mujeres gestantes, seleccionadas de forma aleatoria, en una IPS en el periodo comprendido entre Enero-Julio 2016. El protocolo fue aprobado por el comité de investigación de la IUCMA y por la IPS. Criterio de inclusión: ser gestante entre la semana 35-37 y declaración voluntaria de participación en el estudio mediante la firma del consentimiento informado y de exclusión el uso de antibióticos.
Toma, remisión y procesamiento de las muestras: a las pacientes incluidas, se les tomó muestra con hisopo del introito vaginal y de la región anal. Las muestras se procesaron simultáneamente para qPCR, cultivo en caldo selectivo con posterior siembra en agar sangre de carnero y medio cromogénico para s.agalactiae STRB (ChromIDTMStrepto,BioMérieuxSA.)5. Las colonias se identificaron mediante SISTEMA VITEK 2 (BioMérieux SA).
Extracción y cuantificación del ADN: Se realizó a partir de la muestra tomada con hisopo sumergida en 1 mL de solución salina, empleando el kit comercial de Promega»Wizard(r)Genomic DNA Purification Kit catA1120″, siguiendo las instrucciones del fabricante. El ADN se cuantifico utilizando nanodrop 2000.
Reacción en cadena de la polimerasa en tiempo real: Para la qPCR se utilizaron los iniciadores reportados por Bergsengy cols. 20077, las condiciones óptimas de amplificación fueron determinadas mediante PCR convencional a diferentes concentraciones de MgCl2 (2.5mM-5mM) y a temperaturas entre 50°C-55°C. La qPCR se realizó en el equipo BIORADCFX96REALTIMESISTEM C1000, con el siguiente ciclo térmico: desnaturalización a 95°C/30seg, alineamiento a 55°C/25seg y extensión final a 72°C/20seg por 39 ciclos y una curva de melting 65ºC 0:05 A 95ºC.
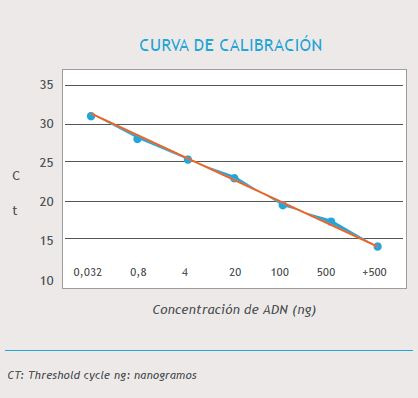
Estandarización, validación e implementación de qPCR: Se realizó curva de calibración empleando diferentes concentraciones de ADN de un aislamiento de S. agalactiae. Los ensayos se realizaron por triplicado. Se determinó la media, coeficiente de variación y desviación estándar. Se definió el límite de detección de DNA en 0,8 ng que corresponde a un tresthold-cicle de 28. (Figura 1) A todos productos de amplificación obtenidos por qPCR se les realizó una electroforesis convencional para confirmar la amplificación del gen blanco. La presencia de inhibidores se evaluó mediante una PCR convencional adicionando ADN de la cepa control al ADN extraído de las muestras de las pacientes.
Figura 1. Curva de calibración CT: Threshold cycle ng: nanogramos

Análisis de la información: La información fue procesada en SPSS22 (IBM, EEUU). A las variables cualitativas se les determinó frecuencias absolutas y relativas, y a las cuantitativas la media, rango y desviación estándar. Para la comparación del agar cromogénico y la qPCR contra la prueba de oro se determinó la sensibilidad, especificidad, valores predictivo positivo y negativo y la precisión diagnostica, con su IC95%.
Consideraciones éticas: El proyecto fue aprobado por Comité de investigaciones de la IUCMA, garantizando el cumplimiento de los lineamientos éticos.
Resultados
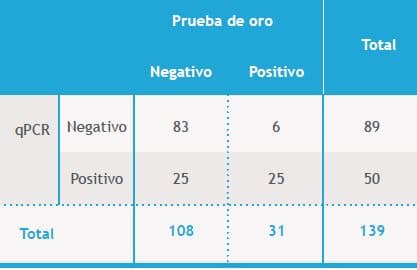
De las 150 muestras tomadas, 139 fueron incluidas en el estudio. La edad promedio de las pacientes fue 26 años, (DS5,8). El 92,4% de las pacientes eran caucásicas, el 73,3% pertenecía a los niveles socioeconómicos 2 y 3, el número de embarazos de las pacientes entre 1- 4. Al evaluar los resultados con relación a estas características, observamos que la prevalencia no presentó diferencias estadísticamente significativas. La prevalencia de colonización por S. agalactiae en las gestantes fue de 20,9%(29 Muestras por prueba de oro, 22,3%(31muestras) por agar cromogénico STRB y 36% (50 Muestras) por qPCR (Tabla1).
Tabla 1. Colonización por Streptococcus agalactiae por los tres métodos evaluados
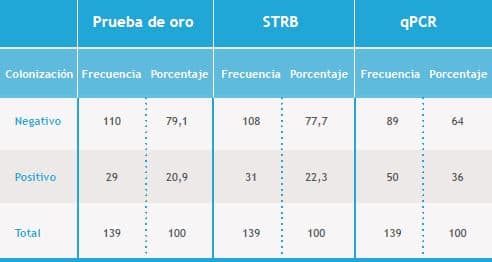
STRB: Medio cromogénico selectivo para screening de Streptococcus agalactiae.
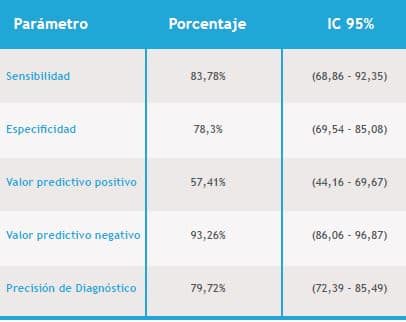
Los resultados encontrados al comparar la qPCR con la prueba de oro fueron: sensibilidad 79,31% (IC del 95%:0,61-0,90) , especificidad 75,45% (IC del 95%:0,66-0,82), valor predictivo positivo 46% (IC del 95%:0,32-0,59) y negativo 93,2% (IC del 95%: 0,86-0,96) tabla 2- tabla 3. Al comparar las pruebas diagnósticas qPCR y cultivo en agar cromogénico: sensibilidad 83,78% (IC: 95%: 0,69-0,92), especificidad 78,3% (IC: 95%:0,69-0,85), valor predictivo positivo 57,41% (IC: 95%:0,440,69 ) y negativo 93,2% (IC: 95%: 0,86-0,96) tabla4 – tabla 5.
Tabla 2. Colonización por Strepotococcus agalactiae empleando qPCR y la prueba de oro
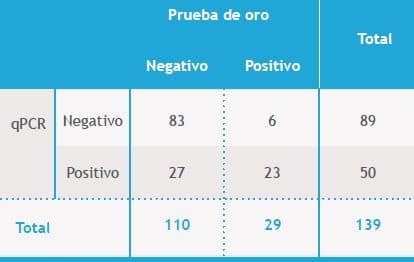
Tabla 3. Rendimiento de qPCR como prueba diagnóstica para detectar Streptococcus agalactiae en mujeres embarazadas en comparación con cultivo como prueba de oro
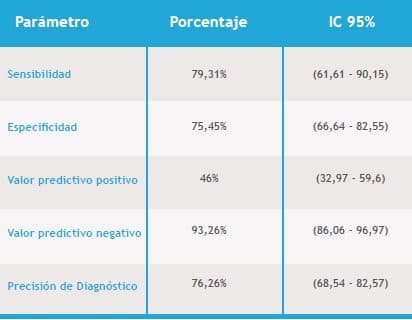
Tabla 4. Colonización por Streptococcus agalactiae empleando qPCR y agar cromogénico STRB
STRB: Medio cromogénico selectivo para screening de Streptococcus agalactiae.
Tabla 5. Rendimiento de qPCR como prueba diagnóstica para detectar Streptococcus agalactiae en mujeres embarazadas en comparación con cultivo en agar cromogénico STRB
Discusión
El empleo de la qPCR permitió aumentar la sensibilidad y la oportunidad diagnostica (El tiempo requerido empleando el cultivo fue de 24-48 horas y por qPCR 6 horas), lo cual impacta en la reducción de riesgos de transmisión neonatal de S. agalactiae, en las complicaciones clínicas de las gestantes y en la mortalidad de los neonatos, lo cual podría representar una disminución en días de estancia y costos hospitalarios por una infección prevenible.
Bergseng y col en 2007, reportaron una prevalencia de colonización del 35%, con sensibilidad de la qPCR frente al cultivo de 97% y especificidad del 99%11, resultados similares empleando el mismo blanco molecular (gen sip), han sido encontrados en nuestro estudio (36%). Feuerschuette y col en 2012 reportaron una prevalencia de 38,2% (21). Nabil Abdullah El Aila, en 2011 empleando los genes sip y cfb mediante qPCR directamente de las muestras, encontraron 27% de positividad frente a 22% del cultivo, una sensibilidad de 95,4%, especificidad de 84,6%, valor predictivo positivo de 63,6% y negativo de 98,5% 10.
Los resultados encontrados en el presente estudio al comparar la qPCR con la prueba de oro y con el cultivo en agar cromogénico, evidencian que la qPCR presentó una mejor sensibilidad y valor predictivo positivo al ser comparado con los resultados obtenidos empleando al agar cromogenico. Los resultados del presente estudio están de acuerdo con los reportados en la literatura; en estos se reportan valores que oscilan entre el 45% y el 99% para sensibilidad y 64,5 a 99,1% para especificidad (21), sin embargo se deben buscar blancos moleculares complementarios para aumentar la sensibilidad diagnóstica y su aplicación de rutina.
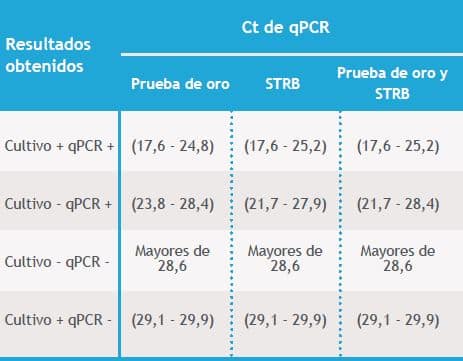
Los 27 resultados positivos por qPCR con resultado negativo en cultivo, pueden explicarse por la presencia de cepas de S. agalactiae no viables o con un número de unidades de colonia muy bajo que no crecen, incluso si están en un medio adecuado. Estas cepas pueden detectarse utilizando qPCR debido a la mayor sensibilidad de este ensayo 24,21, la anterior afirmación se pudo confirmar ya que de los 27 cultivos negativos por la prueba de oro, 4 (14,8%) fueron positivos empleando el cultivo en agar cromogénico STRB. adicionalmente, tal como lo plantean El Aila N.A. et al, la menor sensibilidad del cultivo podría explicarse por la presencia de microorganismos antagonistas como Enterococcus, que inhiben el desarrollo de S. agalactiae y los organismos no viables que pueden ser detectados por PCR pero no así por cultivo 10. Los 6 resultados positivos por cultivo y negativos por qPCR (CT29,1-29,9), (tabla 6) pueden explicarse por la presencia de inhibidores de DNA en la muestra, tal como lo plantea Feuerschuette 201221, dichos inhibidores pueden ser restos de sangre, cremas vaginales entre otros.
Tabla 6. Rango de los valores de Ct en las muestras evaluadas
Se requieren metodologías diagnósticas que permitan detectar la real prevalencia de colonización por S. agalactiae en nuestro medio, por lo que consideramos esencial continuar evaluando diferentes blancos moleculares como los genes cfb, cylE y scpB. Con los resultados de este estudio, podemos concluir que la qPCR aumenta la oportunidad diagnóstica y la sensibilidad en la detección de S. agalactiae en muestras de introito vaginal y región perianal.
Referencias Bibliográficas
1. Di Bartolomeo S, Gentile M, Priore G, Valle S, Di Bella A. Streptococcus agalactiae en embarazadas: Prevalencia en el Hospital Nacional Alejandro Posadas. Rev Argent Microbiol. 2005 Jul/Sep; 37(3):142-44.
2. Guzmán D, Abarzua F, Belmar C, Garcia P. Resultados de la aplicación del protocolo basado en screening para la búsqueda de Streptococcus agalactiae en el tercer trimestre del embarazo: Posible impacto sobre la sepsis neonatal precoz por este agente. Rev chil infectol.2001;18(3):187-92.
3. Cortes H. Prevención de la infección neonatal por estreptococo del grupo B ,¿Es necesaria en nuestro medio?. Rev Colomb Obstet Ginecol. 2005 Jul/Sep; 56(3):152-55.
4. Davies H, Miller M, Faro S, Gregson D, Kehl S, Jordan J. Multicenter study of a rapid molecular-based assay for the diagnosis of group B Streptococcus colonization in pregnant women. Clin. Infect. Dis. 2004; 39:1 9-35.
5. Centers for Disease Control and Prevention (CDC). Prevention of Perinatal Group B Streptococcal Disease Revised Guidelines from CDC. 2010; Vol 59(N° RR – 10): pp. 1 – 23.
6. Ke D, Menard C, Picard FJ, Boissinot M, Ouellette M, Roy PH, et al Development of conventional and real-time PCR assays for the rapid detection of group Streptococci. Clin Chem. 2000;46:324-31.
7. Dmitriev A, Suvorov A, Shen AD, Yang YH. Clinical Diagnosis of Group B Streptococci by scpB gene Based PCR. Indian J Med Res. 2004;119:233-6.
8. Shabayek S, Abdalla S, Abouzeid MH. Comparison of scpB geneand cfb gene polymerase chain reaction assays with culture on Islam medium to detect Group B Streptococcus in pregnancy. Indian J Med Microbiol. 2010;28:320-5.
9. Rasoul Y M, et al. Direct Identification of Streptococcus agalactiae in Vaginal Colonization in Pregnant Women Using Polymerase Chain Reaction. J Compr Ped. 2014 December; 5(4): 233-39.
10. El Aila NA, Tency I, Claeys G, Verstraelen H, Deschaght P, DecatComparison of culture with two different qPCR assays for detection of rectovaginal carriage of Streptococcus agalactiae (group B streptococci). Research in Microbiology. 2011; 162 499-505.
11. Bergseng H, Bevanger L, Rygg M, Bergh K. Real-time PCR targeting the sip gene for detection of group B Streptococcus colonization in pregnant women at delivery. J Med Microbiol. 2007: 56, 223-28.
12. Alfa MJ, et al. Real-time PCR assay provides reliable assessment of intrapartum carriage of group B Streptococcus. J Clin Microbiol. 2010 Sep;48(9):3095-9.
13. Shi CY et al. Detection of maternal colonization of group B Streptococcus in late pregnancy by real-time polymerase chain reaction and its effect on perinatal outcome. Zhonghua Fu Chan KeZaZhi. 2010 Jan;45(1):12-6.
14. Atkins KL, et al. Evaluation of polymerase chain reaction for group B Streptococcus detection using an improved culture method. Obstet Gynecol. 2006 Sep;108:488-91.
15. Rallu F, et al. Sensitivities of Antigen Detection and PCR Assays Greatly Increased Compared to That of the Standard Culture Method for Screening for Group B Streptococcus Carriage in Pregnant Women. J Clin Microbiol. 2006 Mar; 44(3)725-72.
16. Abarzua F. Prevalencia de colonización por streptococcus agalactiae (grupo b) en el tercer trimestre del embarazo. Evaluación del cultivo selectivo. Experiencia en 2192 pacientes. Rev chi lobstet ginecol. 2002;67(2): 89-93.
17. Gavino M, et al. A comparison of a new rapid real-time polymerase chain reaction system to traditional culture in determining group B Streptococcus colonization. Am J Obstet Gynecol.2007 Oct;197(4):388.e1-4.
18. Young BC, et al. Evaluation of a rapid, real-time intrapartum group B Streptococcus assay. Am J Obstet Gynecol.2011 Oct2054372.e1-6
19. Verani JR, McGee L, Schrag SJ. Prevention of perinatal group B streptococcal disease–revised guidelines from CDC, 2010.MMWR Recomm Rep. 2010;59(RR-10):1-36.
20. Laczeski M, Pegels E, Oviedo P, Quiroga M, Vergara M. Molecular profile sand antimicrobial susceptibility of first isolates of Streptococcus agalactiae serotype IX in Argentina. Adv Microbiol.2014 ; 4:317-23.
21. Feuerschuette OM, et al. Performance of RT-PCR in the detection of Streptococcus agalactiae in the anogenital tract of pregnant women. Arch Gynecol Obstet. 2012;286(6):1437-42.
22. Park JS, et al. Usefulness of a Rapid Real-time PCR Assay in Prenatal Screening for Group B Streptococcus Colonization. Ann Lab Med. 2013 Jan;33(1):39-44.
23. K Tanaka et al. Intrapartum group B Streptococcus screening using real time polymerase chain reaction in Japanese population. J Matern Fetal Neonatal Med. 2015 Jan. Pages 130-34.
24. Goodrich J, Miller M. Comparison of culture and 2 real time polymerase chain reaction assays to detect group B Streptococcus during antepartum screening. Diagn Microbiol Infect Dis. 2007; 59:17-2.







