
Detección molecular de patógenos en deposiciones en niños consultantes por diarrea aguda en Servicios de Urgencia
Molecular detection of pathogens in stool samples of children consulting for acute diarrhea in emergency services
Claudia Contreras B.1, Patricia García C.2, Jaime Cerda L.3 y Katia Abarca V.1,4
1División de Pediatría, Facultad de Medicina, Pontificia Universidad Católica de Chile. Santiago, Chile.
2Departamento de Laboratorios Clínicos, Escuela de Medicina. Pontificia Universidad Católica de Chile. Santiago, Chile.
3Departamento de Salud Pública, Facultad de Medicina, Pontificia Universidad Católica de Chile. Santiago, Chile.
4Departamento de Enfermedades Infecciosas e Inmunología Pediátricas, División de Pediatría, Facultad de Medicina, Pontificia Universidad Católica de Chile. Santiago, Chile.
Conflictos de interés: No hubo
Fuente de financiamiento: sin financiamiento
Recibido: 13 de abril de 2023 / Aceptado: 4 de agosto de 2023
Resumen
Introducción: La diarrea aguda continúa siendo una de las principales causas de morbilidad en los niños; Sin embargo, el diagnóstico etiológico presenta limitaciones dada la baja sensibilidad de los métodos tradicionales.
Objetivo: Describir los microorganismos identificados en niños que acudieron al Servicio de Urgencia (SU) de un hospital universitario en Santiago, Chile, por diarrea aguda y a los que se le solicitó panel molecular gastrointestinal.
Métodos: Se revisaron fichas clínicas y resultados de panel gastrointestinal realizados entre junio de 2017 y marzo de 2020.
Resultados: Se incluyeron 198 pacientes, edad promedio de 54,5 meses y 60,6% (120/198) de sexo masculino. La positividad del panel fue de 78,8% (156/198) con 35,3% (55/156) de las muestras polimicrobianas. Se identificaron 229 microorganismos, de los cuales el 72,9% (167/229) corresponden a bacterias, el 25,8% (59/229) a virus y el 1,3% (3/229) a parásitos. Destacaron Campylobacter spp. y Escherichia coli enteropatógena (ECEP) como las bacterias más frecuentemente identificadas. Los pacientes con detección de Campylobacter spp. presentó con mayor frecuencia fiebre (p = 0,00). ECEP se aisló principalmente (82,5%) en muestras polimicrobianas.
Discusión: Los resultados enfatizan el potencial que poseen los estudios moleculares para mejorar el diagnóstico etiológico de la diarrea, pero a la vez llevan a cuestionar el papel patogénico de algunos microorganismos identificados.
Palabras clave: diarrea, etiología, servicios de urgencia, detección por RPC múltiple
Abstract
Background: Acute diarrhea continues to be one of the main causes of morbidity in children, however the etiologica diagnosis presents limitations given the low sensitivity of traditional methods.
Aim: To describe the microorganisms identified in children who attended the emergency department (ED) in Santiago, Chile, due to acute diarrhea and to whom a gastrointestinal panel was requested as part of their study.
Material and Methods: Clinical records and results of the gastrointestinal panel carried out between June 2017 and March 2020 were reviewed.
Results: 198 patients were included, the average age was 54.5 months and 60.6% (120/198) were males. Positivity was 78.8% (156/198) with 35.3% (55/156) of the samples being polymicrobial. 229 microorganisms were identified, of which 72.9% (167/229) corresponded to bacteria, 25.8% (59/229) to viruses, and 1.3% (3/229) to parasites. Campylobacter spp. and enteropathogenic Escherichia coli (EPEC) were the most frequently identified bacteria. Patients with detection of Campylobacter spp. presented a higher frequency of fever (p = 0.00). EPEC was isolated in 82.5% of the cases in polymicrobial samples.
Discussion: The results emphasize the potential of molecular studies to improve the etiological diagnosis of diarrhea and at the same time lead to question the pathogenic role of some microorganisms.
Keywords: diarrhea, etiology, emergency departments, multiplex PCR detection
Introducción
Pese a importantes avances mundiales en las condiciones sanitarias, la diarrea continúa siendo una de las principales causas de morbilidad en niños menores de 5 años de edad. Anualmente, se producen entre 1,1 y 1,7 millones de episodios de diarrea y entre 450.000 y 525.000 muertes por esta causa1, 2.
La definición de diarrea aguda incluye la presencia de tres o más episodios de deposiciones líquidas en 24 horas, con menos de 14 días de duración1. La etiología infecciosa predomina mundialmente, siendo el rotavirus el principal agente aislado y aquel con la mayor mortalidad asociada2. Sin embargo, los agentes infecciosos varían de acuerdo con las condiciones socioeconómicas y sanitarias, así como también según la edad del paciente. En Chile, clásicamente se ha descrito un predominio de virus por sobre bacterias y parásitos, siendo identificados rotavirus y norovirus como las principales causas de diarrea aguda infecciosa en niños menores de 5 años3,4.
El diagnóstico etiológico presenta limitaciones dada la baja sensibilidad de los métodos tradicionales y el amplio número de patógenos asociados5,6. En este contexto, surgen nuevos métodos que apoyan el diagnóstico etiológico con el uso de técnicas moleculares. Dentro de ellos, se encuentra el uso del panel gastrointestinal (GI) ampliado para la detección de 22 patógenos por técnica de reacción de polimerasa en cadena (RPC). Este último destaca por su alta sensibilidad y especificidad para detectar, tanto bacterias como virus y parásitos6. Lo anterior ha permitido un importante avance en el estudio etiológico del síndrome diarreico entregando información de gran valor epidemiológico. Además, se ha descrito un impacto clínico, principalmente con relación a la toma de decisiones terapéuticas por parte del personal de la salud, volviéndose más rápidas y dirigidas al agente identificado7. Por último, su uso ha dejado en evidencia la alta tasa de coinfecciones y el importante porcentaje de Escherichia coli diarreogénicas como agentes identificados 5,8. Sin embargo, al identificarse el material genético de los distintos microorganismos, estas técnicas no permiten asignar causalidad.
En Chile, existen escasos estudios que involucran el uso de esta herramienta de tecnología avanzada, especialmente en población pediátrica. En el año 2016, Farfán y cols. Publicaron su experiencia preliminar con el uso del panel GI Film Array® en una clínica privada para la detección de patógenos entéricos en deposiciones, analizando 305 muestras de adultos y niños, reportando una positividad de 67,5%, con predominio de Escherichia coli enteropatógena (ECEP), norovirus y Giardia lamblia para los patógenos bacterianos, virales y parasitarios, respectivamente5. Posteriormente, Valenzuela y cols. realizó un estudio prospectivo en población adulta chilena que acudió al Servicio de Urgencia de nuestra institución con el objetivo de caracterizar la etiología de la diarrea aguda y evaluar su evaluación clínica, usando el panel GI FilmArray®. Dicho estudio reportó como agentes más frecuentes ECEP, Campylobacter spp y E. coli enteroagregativa (ECEA)8. En vista del importante valor epidemiológico de la información obtenida mediante esta técnica molecular y la ausencia de estudios en población pediátrica en Chile, el objetivo de este estudio fue la identificación de microorganismos en heces de niños que consultaron por diarrea aguda en servicios de urgencia de la Red Salud UC Christus, mediante panel molecular GI.
Materiales y Métodos
Se realizó un estudio descriptivo y retrospectivo que incluyó la revisión de los resultados de los paneles GI (Biofire® FilmArray®) en niños que consultaron por diarrea aguda en los Servicios de Urgencia del Hospital Clínico de la Pontificia Universidad Católica y de la Clínica San Carlos de Apoquindo, desde junio del año 2017, fecha en que se implementó esta prestación, hasta marzo del año 2020.
Como criterio de inclusión se mostraron pacientes menores de 16 años de edad, con un cuadro clínico caracterizado por diarrea de menos de 14 días de evolución y que contaran con el resultado de un panel GI solicitado en su consulta al Servicio de Urgencia. No hubo criterios de exclusión. Considerando la naturaleza retrospectiva del estudio, dado que los participantes no fueron contactados y que se manejaron los datos asignando un código a cada caso, se obtuvo dispensa de solicitud de Consentimiento Informado por el Comité Ético Científico de Ciencias de la Salud de la PUC (ID del estudio 210107004, Acta de aprobación del 11 de marzo de 2021). Se registró en una base de datos el resultado del panel GI y la información epidemiológica y clínica extraída de la ficha electrónica de urgencia de cada paciente.
Además, se revisaron las epicrisis en los casos que fueron hospitalizados y de las atenciones ambulatorias previas y posteriores a la consulta, cuando estas se habían realizado en la red de salud.
Para el análisis de los datos, se utilizó estadística descriptiva y para la comparación de variables categóricas se utilizó la prueba exacta de Fisher, considerando estadísticamente significativo un valor p < 0,05. Los cálculos fueron realizados utilizando la plataforma OpenEpi Versión 3.01.
Resultados
Se revisaron las fichas clínicas de urgencia de 250 pacientes en que el panel GI fue solicitado en el período de estudio, de los cuales 198 fueron incluidos. Se excluiron nueve pacientes por no contar con datos suficientes que permitan definir la duración del cuadro o los síntomas presentados, siete pacientes por no presentar diarrea y 36 pacientes por presentar diarrea de 14 o más días de duración.
En relación a las características epidemiológicas de la población estudiada, la edad promedio fue de 54,5 meses (DE 48,5 meses) y la mediana 37,9 meses (rango intercuartil 61,6 meses), con 60,6% (120 /198) de los pacientes de sexo masculino.
Respecto a los resultados del panel GI, se observó una positividad de 78,8% (156/198), con 229 microorganismos identificados. Del total de muestras positivas, 64,7% (101/156) resultaron monomicrobianas y 35,3% (55/156) polimicrobianas: se identificaon dos microorganismos en 25,6% (40/156), tres microorganismos en 7,7 % (12/156) y cuatro microorganismos en 1,9% (3/156).
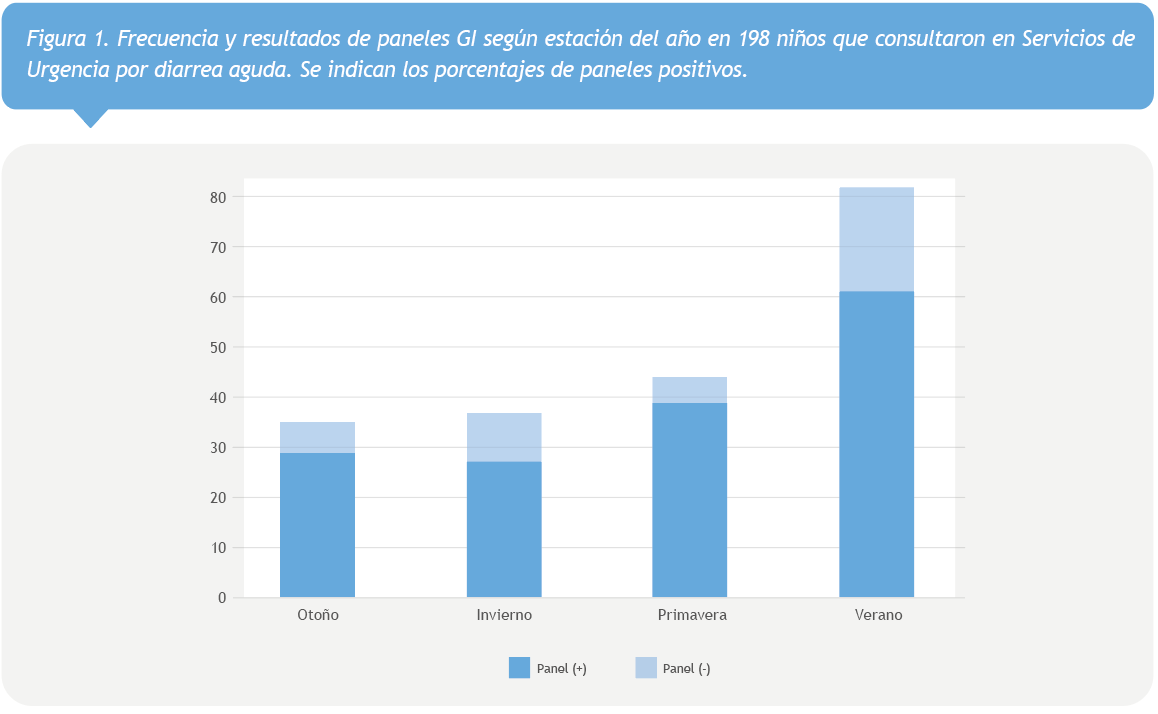
La Figura 1 muestra la positividad del examen en relación a las estaciones del año. Pese a que se observara una tendencia a mayor positividad en los meses de verano, no existieron diferencias estadísticamente significativas al comparar los resultados de cada estación entre sí.
De los 229 microorganismos identificados, 72,9% (167/229) correspondieron a bacterias, 25,8% (59/229) a virus y 1,3% (3/229) a parásitos. En la Tabla 1 se describen los agentes infecciosos identificados, así como el número identificado de este agente en muestras polimicrobianas.
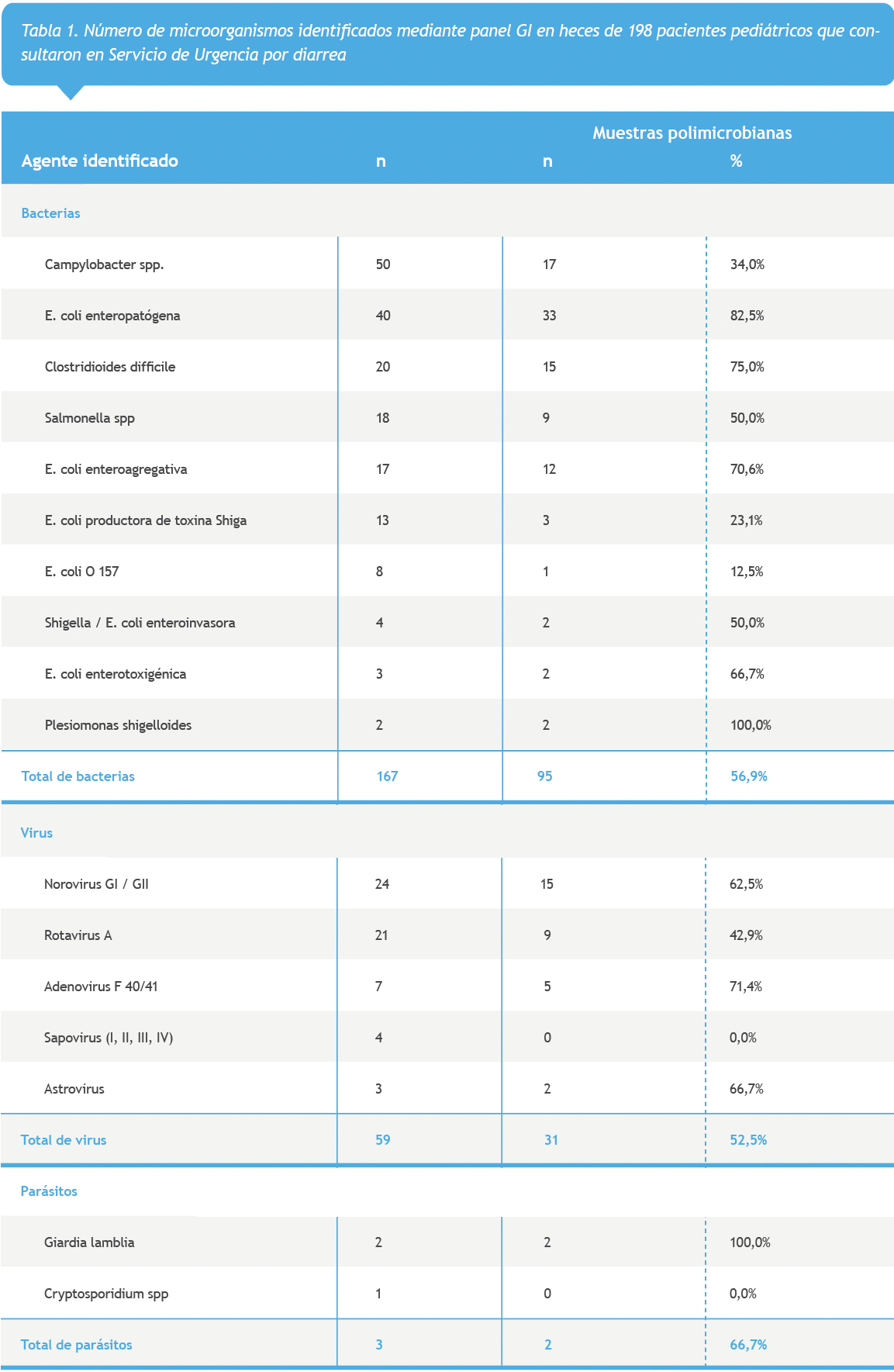
Cabe destacar la alta tasa de detección de bacterias, destacando Campylobacter spp (que puede incluir a las especies C. jejuni, C. coli y C. upsaliensis) y ECEP como las especies más frecuentemente identificadas. Asimismo, destaca la alta tasa de detección de microorganismos en muestras polimicrobianas, tales como ECEP, Clostridioides difficile y ECEA. No se detectaron Cyclospora cayetanensis, Entamoeba histolytica, Vibrio (V. cholerae, V. parahaemolyticus, V. vulnificus) ni Yersinia enterocolitica.
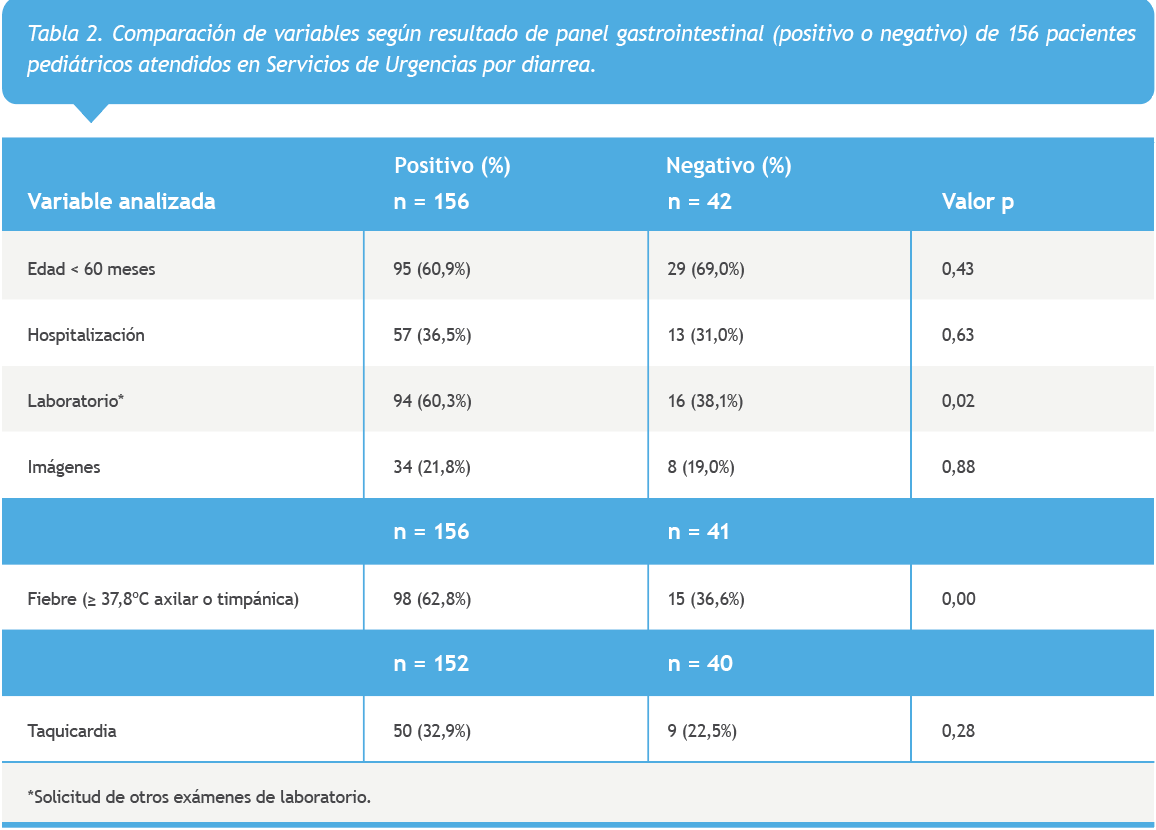
Las Tablas 2 y 3 comparan variables epidemiológicas ( edad), clínicas (fiebre y taquicardia en signos vitales) y del manejo (hospitalización y solicitud de otros exámenes de laboratorio o imágenes) de los pacientes, según el resultado positivo o negativo y monomicrobiano o polimicrobiano del panel GI, respectivamente. Destaca el mayor porcentaje de solicitud de solicitudes de laboratorio (60,3 vs 38,1%) y fiebre (62,8 vs 36,6%) en el grupo con panel GI positivo y el mayor porcentaje de pacientes menores de 60 meses de edad con resultado de panel GI polimicrobiano (81,0 vs 49,5%).
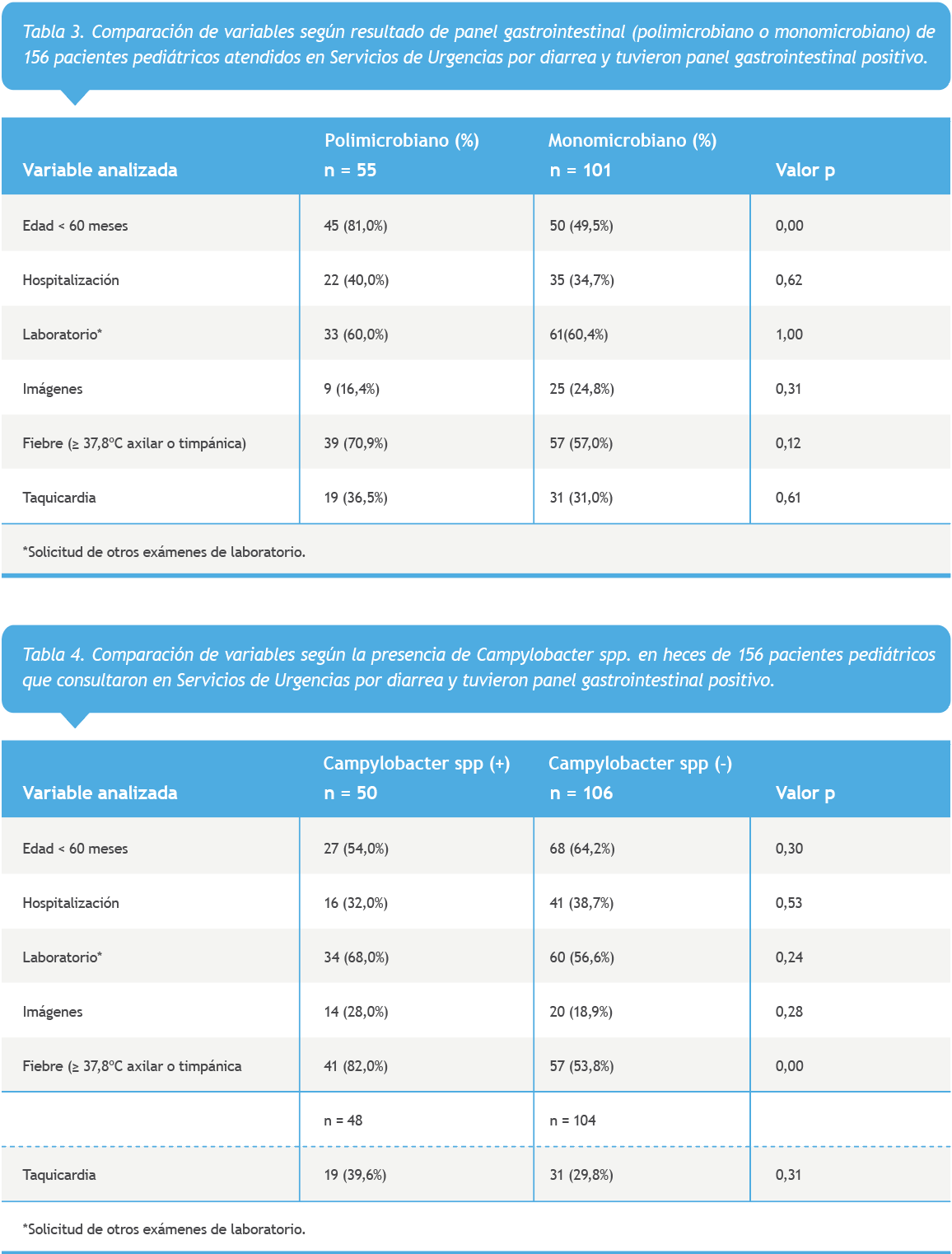
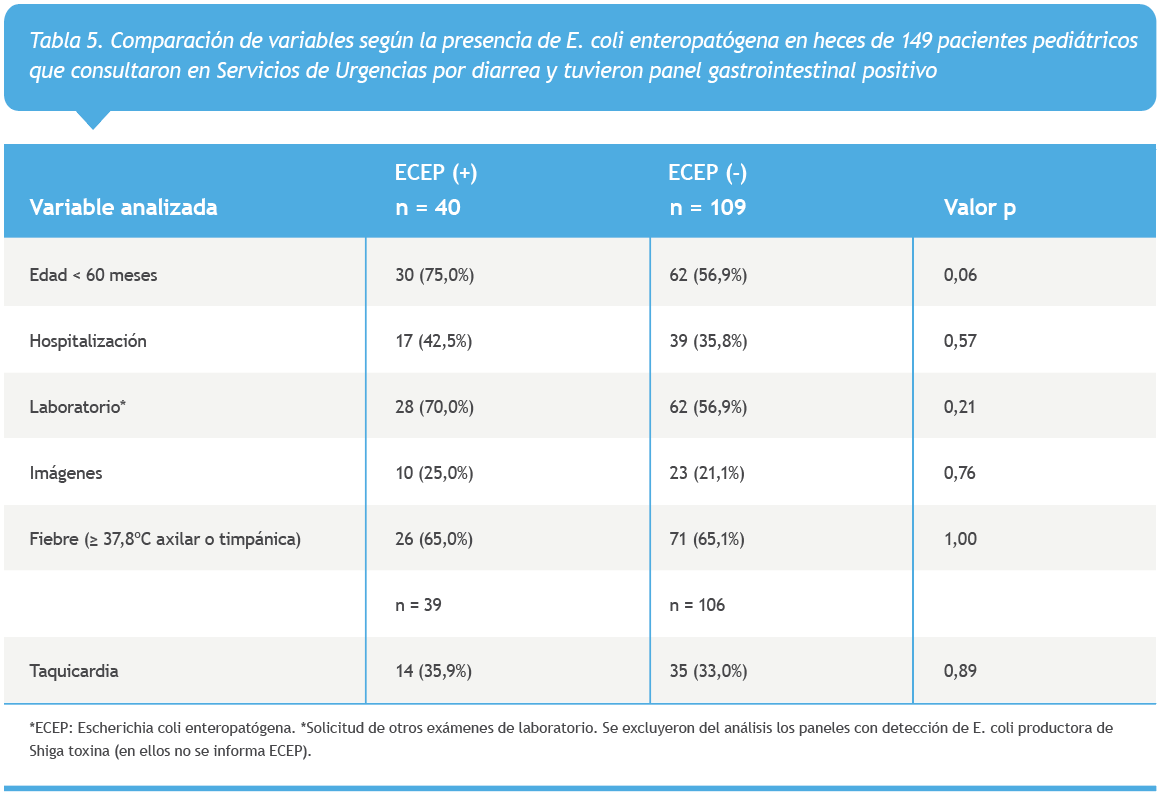
Por otro lado, las Tablas 4 y 5 comparan estas variables según detección de los agentes más frecuentemente identificados, Campylobacter spp. y ECEP. Destaca la mayor frecuencia de fiebre (82,0 vs 53,8%) en aquellos pacientes en los que se detectó Campylobacter spp. (p = 0,00).
Discusión
En este estudio, la positividad de las muestras analizadas alcanzó un 78,8% (156/198), cifra elevada en comparación con la tasa de detección del FilmArray®. Este amplio rango de detección probablemente se encuentra en el contexto de las distintas poblaciones analizadas: adultos versus niños, y la procedencia de las muestras: ambulatorio, hospitalizado o servicio de urgencia. Así, al revisar aquellos estudios en que se detalla el ámbito de la salud desde donde se obtuvo la muestra a analizar, se observa que la positividad se eleva hasta 68% cuando se trata de resultados obtenidos desde los servicios de urgencia13–9 IG publicada en la literatura médica, la que varía entre 33 y 54,2%,10. Al comparar nuestros resultados con aquellos obtenidos en el estudio de Valenzuela y cols. en población adulta chilena que acudió al Servicio de Urgencia de nuestra institución, se observa que la positividad del panel GI es mayor en la población pediátrica de nuestro estudio (78,8 vs 66,8%, valor p = 0,01).
La detección de bacterias predominó por sobre virus y parásitos, constituyendo estas el 72,9% (167/229) del total de microorganismos detectados. Lo anterior podría explicarse dado que el estudio de panel GI corresponde a un examen de elevado costo en nuestro país, que por lo general se reserva para diarreas de mayor gravedad y/o con presencia de elementos patológicos tales como sangre o mucosidad. Dado el carácter retrospectivo de nuestro estudio, no contamos con la descripción de este último dato en la anamnesis de la totalidad de los pacientes analizados. No obstante, en el 47% de las fichas revisadas (93/198) se registraba la presencia de sangre en las deposiciones. De estos, el 83,9% (78/93) presentó un panel GI con resultado positivo en los que se detectó al menos una bacteria en el 94,9% (74/78).
Dentro de los microrganismos detectados con mayor frecuencia destaca Campylobacter spp y E. coli diarreogénicas, principalmente ECEP, similar a lo observado en el estudio local de Valenzuela y cols.8. En el ámbito internacional, otros estudios que han utilizado el panel GI (FilmArray®) en la búsqueda etiológica muestran resultados similares, tal como un estudio multicéntrico realizado en Europa con más de 700 muestras de declaraciones analizadas9. Esto difiere parcialmente de los hallazgos en publicaciones de Estados Unidos de Norteamérica, donde se observa C. difficile y E. coli diarreogénicas con mayor frecuencia y un bajo porcentaje de detección de Campylobacter spp.6,10.
Respecto a Campylobacter spp., destaca que se encontró como patógeno único en aproximadamente dos de cada tres muestras en las que se le identificó. Además, se detectó este microorganismo en el 52,6% (41/78) del total de pacientes en los que se logró rescatar el antecedente de sangre en deposiciones y que contaban con un panel GI positivo. Lo anterior, sumado a la alta frecuencia de Campylobacter spp. en las muestras analizadas y la mayor frecuencia de fiebre en los pacientes que contaban con este microorganismo aislado en deposiciones, da cuenta de la importancia de la búsqueda dirigida de este microorganismo en el estudio etiológico de pacientes con diagnóstico de síndrome diarreico agudo, especialmente aquellos que presenten disentería. Como una alternativa de menor costo, se puede considerar solicitar el cultivo de Campylobacter. Por otro lado, ECEP es una causa conocida, tanto de diarrea aguda como diarrea persistente en niños14 y en nuestro estudio destacó como el segundo agente infeccioso más frecuente. , aunque en solo el 17,5% de las muestras fueron encontradas como patógeno único. Estudios diversos han encontrado este microorganismo con una frecuencia similar en pacientes con diarrea y en controles15–17
Con relación a C. difficile, correspondió al 8,7% (20/229) del total de microorganismos detectados, ocupando el tercer lugar en frecuencia dentro de las bacterias detectadas. Cabe destacar que tres de cada cuatro muestras positivas para C. difficile resultaron polimicrobianas y 60% (12/20) fueron obtenidos de niños bajos 2 años de edad. Esto coincide con lo descrito en la literatura médica, donde se cuestiona el papel patogénico de este microorganismo, considerando que hasta el 70% de los niños bajo un año de edad podrían estar colonizados18. . Más aún, la Academia Americana de Pediatría recomienda limitar las pruebas de detección para este microorganismo en lactantes bajo un año de edad, a aquellos pacientes con enfermedad de Hirschsprung, trastornos de la motilidad intestinal, o frente a un brote epidémico. Además, menciona que resulta difícil interpretar resultados positivos en aquellos pacientes menores de 3 años19.
El laboratorio de la Red de Salud UC Christus diferencia la identificación de bacterias en cultivo de deposiciones de acuerdo a la edad del paciente: en pacientes menores de 10 años de edad se realiza la búsqueda de tres bacterias; mientras que en los pacientes mayores de 10 años solo se buscan las dos último. Cabe destacar que, si bien la detección de bacterias en nuestro estudio fue elevada, en solo 18% (30/167) fueron las bacterias antes mencionadas, lo que deja en evidencia el bajo porcentaje de diagnóstico etiológico al que podemos llegar con el uso de los métodos tradicionales., Salmonella y Shigella, O:157
Se detectó E. coli 0:157 en tan solo ocho pacientes, con edades comprendidas entre 45 y 144 meses (edad promedio 82,72 meses). Solo una de las muestras positivas resultó polimicrobiana. De los ocho pacientes, dos de ellos requirieron hospitalización y uno de ellos evolucionó con síndrome hemolítico urémico requiriendo manejo en una unidad de paciente crítico.
Con relación a la detección de virus, estos dieron cuenta de 25,8% (59/229) del total de microrganismos identificados. Dentro de estos, destacó el norovirus como el agente viral más frecuentemente detectado (24/59). Lo anterior coincide con lo descrito, tanto en estudios nacionales como internacionales, en los que se utilizó el panel GI como método de diagnóstico etiológico8–22,21. Además, se ha descrito una mayor frecuencia de cuadros diarreicos por norovirus en pacientes menores de 5 años21,20 causante de al menos el 95% de los brotes no bacterianos de gastroenteritis, siendo la principal causa de brotes de gastroenteritis aguda en el mundoCaliciviridae,. Norovirus constituye un género de virus ARN dentro de la familia 10
Las muestras en las que se detectó más de un microorganismo ascienden hasta el 35,3% (55/156) del total de paneles GI positivos. Esta cifra se eleva por sobre lo identificado en el estudio de Valenzuela y cols., donde las muestras polimicrobianas representaron el 27,1% (54/199) de las analizadas. Sin embargo, al revisar las variables clínicas y de manejo (Tabla 3), que de manera indirecta podrían orientar en la gravedad de los pacientes, vemos que no hay diferencias estadísticamente significativas entre el grupo de pacientes con muestras polimicrobianas y aquellos con muestras monomicrobianas. Lo anterior lleva a cuestionar el papel patogénico de ciertos microorganismos, especialmente aquellos que se encontraron en muestras polimicrobianas en más del 70% de los casos tales como ECEP, ECEA y C. difícil.
Dentro de las limitaciones de nuestro estudio, destaca que la población estudiada se limitó a aquellos pacientes con panel GI solicitado y, por tanto, estos datos deben interpretarse con precaución ante un probable sesgo de selección dado que este examen se suele solicitar a aquellos pacientes con un cuadro de mayor gravedad y por su elevado costo no es accesible a la totalidad de los casos en que se solicita. Además, cabe destacar que la población estudiada representa a niños que consultan en centros de atención de salud privados por lo que los resultados podrían no ser extrapolables al resto de la población nacional y presentar diferencias según la localización geográfica y nivel socioeconómico de los pacientes.
A modo de conclusión, es posible que, con el advenimiento de técnicas moleculares y la consecuente mayor detección de patógenos, la etiología de la diarrea aguda presente variaciones durante los próximos años.
En nuestro estudio destaca una alta detección de bacterias, incluso por sobre virus, y un número elevado de casos de diarrea causada por Campylobacter spp. , especialmente en aquellos pacientes con disentería, información de gran valor para orientar el estudio etiológico y lograr un inicio de tratamiento oportuno.
Más aún, nuestro estudio deja en evidencia la relevancia de llevar a cabo futuros estudios prospectivos, donde sea posible evaluar con mayor exactitud el rol de ciertos microorganismos en los cuadros de gastroenteritis aguda para evitar atribuir rol patógeno en forma equivocada e iniciar tratamientos innecesarios.
Fuente de financiamiento: sin financiamiento
Referencias Bibliográficas
1 Organización Mundial de la Salud (2 de mayo, 2017), Enfermedad diarreica. https://www.who.int/news-room/fact-sheets/detail/diarrhoealdisease. Accedido: 10 de marzo de 2022. [ Enlaces ]
2 GBD 2016 Colaboradores en Enfermedades Diarreicas. Estimaciones de la morbilidad, mortalidad y etiologías de la diarrea a nivel mundial, regional y nacional en 195 países: un análisis sistemático para el Estudio de carga global de enfermedades 2016. Lancet Infect Dis. 2018; 18(11): 1211-28. doi: 10.1016/S1473-3099(18)30362-1. Publicación electrónica del 19 de septiembre de 2018. https://doi.org/10.1016/S1473-3099(18)30362-1 [ Enlaces ]
3 Yalda Lucero A. Etiología y manejo de la gastroenteritis aguda infecciosa en niños y adultos. Revista Médica Clínica Las Condes, 2014; 25(3): 463-72. https://doi.org/10.1016/S0716-8640(14)70063-X. [ Enlaces ]
4 O’Ryan M, Pérez-Schael I, Mamani N, Peña A, Salinas B, González G, et al. Visitas médicas y hospitalizaciones asociadas a rotavirus en América del Sur: un estudio prospectivo en tres grandes hospitales centinela. Pediatr Infect Dis J. 2001 julio; 20(7): 685-93. doi: 10.1097/00006454-200107000-00009. [ Enlaces ]
5 Farfán M, Piemonte P, Labra Y, Henríquez J, Candía E, Torres J P. Panel Filmarray GI® en la detección de patógenos entéricos en deposiciones: experiencia preliminar Rev Chilena Infectol. 2016; 33(1): 89-91. Español. doi: 10.4067/S0716-10182016000100016. PMID: 26965886. [ Enlaces ]
6 Buss SN, Leber A, Chapin K, Fey P D, Bankowski MJ, Jones M K, et al. Evaluación multicéntrica del panel gastrointestinal BioFire FilmArray para el diagnóstico etiológico de gastroenteritis infecciosa. J Clin Microbiol. 2015; 53(3): 915-25. doi: 10.1128/JCM.02674-14. Publicación electrónica del 14 de enero de 2015. PMID: 25588652; PMCID: PMC4390666. [ Enlaces ]
7 Cybulski R J Jr, Bateman A C, Bourassa L, Bryan A, Beail B, Matsumoto J, Cookson B T, Fang F C. Impacto clínico de un panel de reacción en cadena de la polimerasa gastrointestinal múltiple en pacientes con gastroenteritis aguda. Clin Infect Dis. 2018; 67(11): 1688-96. doi: 10.1093/cid/ciy357. PMID: 29697761. [ Enlaces ]
8 Valenzuela C, Legarraga P, Peña A, Arenas A, Berkowitz L, Ramírez G, et al. Caracterización etiológica y clínica de gastroenteritis adquirida en la comunidad en pacientes adultos en un servicio de urgencias de Chile mediante el panel FilmArray GI. Más uno. 2018; 13(11): e0207850. doi: 10.1371/journal.pone.0207850. PMID: 30475924; PMCID: PMC6258233. [ Enlaces ]
9 Spina A, Kerr KG, Cormican M, Barbut F, Eigentler A, Zerva L, et al. Espectro de enteropatógenos detectados por FilmArray GI Panel en un estudio multicéntrico de gastroenteritis adquirida en la comunidad. Infectación de Clin Microbiol. 2015; 21(8): 719-28. doi: 10.1016/j.cmi.2015.04.007. Publicación electrónica del 20 de abril de 2015. PMID: 25908431. [ Enlaces ]
10 Stockmann C, Pavia A T, Graham B, Vaughn M, Crisp R, Poritz MA, et al. Detección de 23 patógenos gastrointestinales entre niños que presentan diarrea. J Pediátrico Infect Dis Soc. 2017; 6(3): 231-8. doi: 10.1093/jpids/piw020. PMID: 27147712; PMCID: PMC5907859. [ Enlaces ]
11 Buss SN, Leber A, Chapin K, Fey P D, Bankowski MJ, Jones M K, et al. Evaluación multicéntrica del panel gastrointestinal BioFire FilmArray para el diagnóstico etiológico de gastroenteritis infecciosa. J Clin Microbiol. 2015; 53(3): 915-25. doi: 10.1128/JCM.02674-14. Publicación electrónica del 14 de enero de 2015. PMID: 25588652; PMCID: PMC4390666. [ Enlaces ]
12 Khare R, Espy MJ, Cebelinski E, Boxrud D, Sloan LM, Cunningham SA, et al. Evaluación comparativa de dos paneles multiplex comerciales para la detección de patógenos gastrointestinales mediante el uso de muestras clínicas de heces. J Clin Microbiol. 2014;52(10): 3667-73. doi: 10.1128/JCM.01637-14. Publicación electrónica del 6 de agosto de 2014. PMID: 25100818; PMCID: PMC4187753. [ Enlaces ]
13 Murphy C N, Fowler R C, Iwen P C, Fey P D. Evaluación del panel gastrointestinal BioFire FilmArray® en un hospital académico del Medio Oeste. Eur J Clin Microbiol Infect Dis. 2017; 36(4): 747-54. doi: 10.1007/s10096-016-2858-7. Publicación electrónica del 12 de diciembre de 2016. PMID: 27957599. [ Enlaces ]
14 Croxen M A, Law R J, Scholz R, Keeney KM, Wlodarska M, Finlay B B. Avances recientes en la comprensión de los patógenos entéricos Escherichia coli. Clin Microbiol Rev.2013; 26(4): 822-80. doi: 10.1128/CMR.00022-13. PMID: 24092857; PMCID: PMC3811233. [ Enlaces ]
15 Imdad A, Foster M A, Iqbal J, Fonnesbeck C, Payne D C, Zhang C, et al. Escherichia coli diarreógena y gastroenteritis aguda en niños del condado de Davidson, Tennessee, Estados Unidos: un estudio de casos y controles. Pediatr Infect Dis J. 2018; 37(6): 543-548. doi: 10.1097/INF.0000000000001908. PMID: 29341983; PMCID: PMC5962020. [ Enlaces ]
16 Knutton S, Shaw R, Phillips AD, Smith HR, Willshaw GA, Watson P, et al. Análisis fenotípico y genético de Escherichia coli asociada a la diarrea aislada de niños en el Reino Unido. J Pediatr Gastroenterol Nutr. 2001; 33(1): 32-40. doi: 10.1097/00005176-200107000-00006. PMID: 11479405. [ Enlaces ]
17 Ochoa T J, Mercado E H, Durand D, Rivera F P, Mosquito S, Contreras C, et al. Frecuencia y patotipos de Escherichia coli diarrogénicas en niños peruanos con y sin diarrea. Rev Perú Med Exp Salud Pública. 2011; 28(1): 13-20. Español. doi: 10.1590/s1726-46342011000100003. PMID: 21537764. [ Enlaces ]
18 Jangi S, Lamont J T. Colonización asintomática por Clostridium difficile en bebés: implicaciones para la enfermedad en etapas posteriores de la vida. J Pediatr Gastroenterol Nutr. 2010; 51(1): 2-7. doi: 10.1097/MPG.0b013e3181d29767. PMID: 20512057. [ Enlaces ]
19 Schutze GE, Willoughby RE; Comité de Enfermedades Infecciosas; Academia Americana de Pediatría. Clostridium difficile infección en bebés y niños. Pediatría. 2013; 131(1): 196-200. doi: 10.1542/peds.2012-2992. Publicación electrónica del 31 de diciembre de 2012. PMID: 23277317. [ Enlaces ]
20 Karst S M. Patogénesis de norovirus, virus de ARN emergentes. Virus. 2010; 2(3): 748-81. doi: 10.3390/v2030748. Publicación electrónica del 23 de marzo de 2010. PMID: 21994656; PMCID: PMC3185648. [ Enlaces ]
21 Norovirus a nivel mundial, https://www.cdc.gov/norovirus/trends-outbreaks/worldwide.html (Accedido: 7 de marzo de 2023). [ Enlaces ]
22 Grytdal SP, DeBess E, Lee LE, Blythe D, Ryan P, Biggs C, et al. Incidencia de norovirus y otros patógenos virales que causan gastroenteritis aguda (AGE) entre las poblaciones de miembros de Kaiser Permanente en los Estados Unidos, 2012-2013. Más uno. 2016; 11(4): e0148395. doi:10.1371/journal.pone.0148395. PMID: 27115485; PMCID: PMC4846013. [ Enlaces ]
Correspondencia a: Katia Abarca Villaseca kabarca@uc.cl
Conflictos de intereses: No hubo
Este es un artículo de Acceso Abierto distribuido bajo los términos de la Licencia No Comercial de Atribución Creative Commons, que permite el uso, distribución y reproducción no comerciales sin restricciones en cualquier medio, siempre que el trabajo original esté debidamente citado.






